
Spisu treści:
- Krok 1: Skonfiguruj zasobnik Google Cloud Storage
- Krok 2: Sformatuj swoje dane i utwórz zbiór danych Csv
- Krok 3: Prześlij swoje spektrogramy do swojego wiadra
- Krok 4: Prześlij swój zbiór danych CSV
- Krok 5: Utwórz zbiór danych
- Krok 6: Utwórz swój model AutoML
- Krok 7: Przetestuj swój model
- Krok 8: Zainstaluj swój model w ThinkBioT
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:29.
- Ostatnio zmodyfikowany 2025-01-23 15:03.

ThinkBioT został zaprojektowany jako „Plug and Play” z modelami TensorFlow Lite zgodnymi z Edge TPU.
W tej dokumentacji omówimy tworzenie spektrogramów, formatowanie danych i używanie Google AutoML.
Kod w tym samouczku zostanie napisany w bash, więc będzie kompatybilny z wieloma platformami.
Zależności
- Jednak przed rozpoczęciem będziesz musiał zainstalować Sox program audio wiersza poleceń kompatybilny z urządzeniami Windows, Mac i Linux.
- Jeśli korzystasz z urządzenia z systemem Windows, najłatwiejszym sposobem uruchamiania skryptów bash jest Git, więc polecam i pobranie i zainstalowanie tego, ponieważ jest to przydatne na wiele sposobów,
- Do edycji kodu użyj swojego ulubionego edytora lub zainstaluj NotePad ++ dla systemu Windows lub Atom dla innych systemów operacyjnych.
**Jeśli masz istniejący model TensorFlow lub chcesz spróbować przenieść naukę z istniejącym modelem, zapoznaj się z dokumentacją Google Coral.
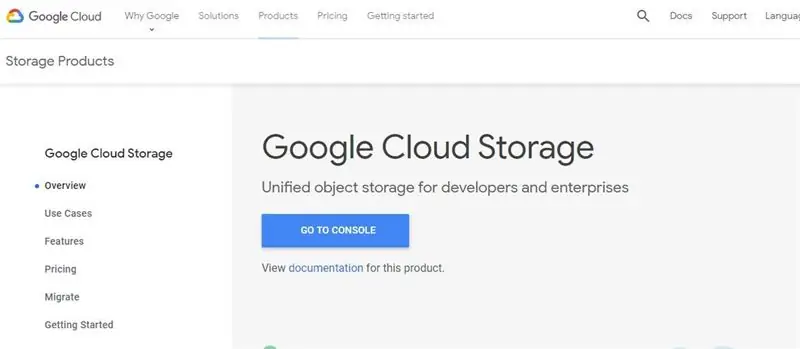
Krok 1: Skonfiguruj zasobnik Google Cloud Storage
1. Zaloguj się na swoje konto Gmail (lub utwórz je, jeśli nie masz konta Google)
2. Przejdź do strony wyboru projektów i utwórz nowy projekt dla swojego modelu i plików spektrogramów. Aby przejść dalej, musisz włączyć rozliczenia.
3. Wejdź na https://cloud.google.com/storage/ i naciśnij przycisk tworzenia zasobnika u góry strony.
4. Wprowadź żądaną nazwę kubełka i utwórz kubeł akceptujący ustawienia domyślne.
Krok 2: Sformatuj swoje dane i utwórz zbiór danych Csv
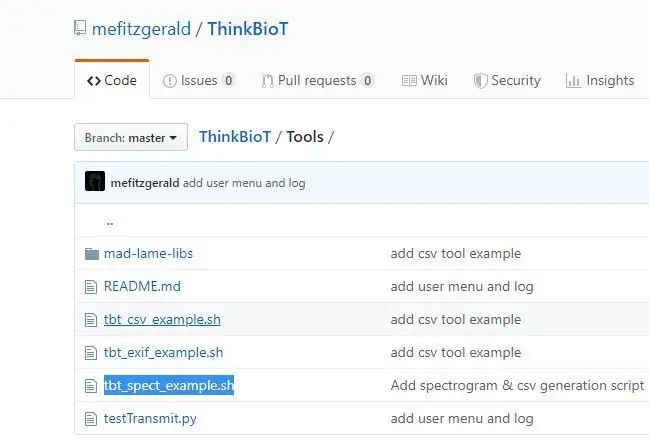
Zaprojektowałem pomocny skrypt do tworzenia pliku dataset.csv potrzebnego do stworzenia modelu. Plik zbioru danych łączy obrazy w zasobniku z ich etykietami w zbiorze danych.
1. Pobierz repozytorium ThinkBioT z GitHub i
2. Skopiuj plik tbt_spect_example.sh z katalogu Tools do nowego folderu na pulpicie.
3. Dodaj pliki audio, których chcesz użyć w swoim modelu, umieszczając je w folderach, które mają swoją etykietę (tj. jak chcesz je posortować. Na przykład, jeśli chcesz zidentyfikować psy lub koty, możesz mieć folder pies, z odgłosami szczekania LUB folder o nazwie kot z odgłosami kota itp.
4. Otwórz plik tbt_spect_example.sh za pomocą Notepad++ i zastąp „yourbucknamename” w wierszu 54. nazwą Twojego zasobnika Google Storage. Na przykład, jeśli Twój wiadro nazywa się myModelBucket, linia zostanie zmieniona na
bucket="gs://myModelBucket/spektro-dane/"
5. Uruchom kod, wpisując następujące polecenie w swoim terminalu Bash, kod uruchomi się i utworzy plik csv z etykietami oraz katalog o nazwie spektro-dane na pulpicie z wynikami spektrogramów.
sh tbt_spect_example.sh
Krok 3: Prześlij swoje spektrogramy do swojego wiadra
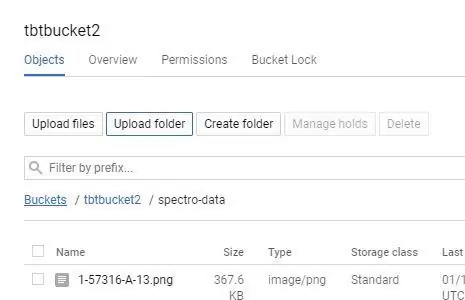
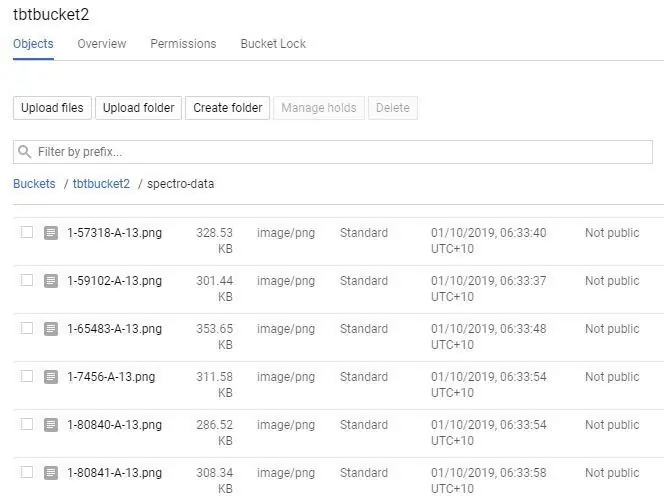
Istnieje kilka sposobów przesyłania do Google Storage, najłatwiejszy to bezpośrednie ładowanie folderu;
1. Kliknij nazwę swojego zasobnika na stronie Google Storage.
2. Wybierz przycisk „UPLOAD FOLDER” i wybierz katalog „spectro-data/” utworzony w ostatnim kroku.
LUB
2. Jeśli masz dużą ilość plików, możesz ręcznie utworzyć katalog "spectro-data/" wybierając "CREATE FOLDER", a następnie przejdź do folderu i wybierz "UPLOAD FILES". Może to być świetna opcja dla dużych zestawów danych, ponieważ możesz przesyłać spektrogramy w sekcjach, nawet używając wielu komputerów, aby zwiększyć prędkość przesyłania.
LUB
2. Jeśli jesteś zaawansowanym użytkownikiem, możesz również przesyłać za pośrednictwem Google Cloud Shell;
gsutil cp dane-spektro/* gs://nazwa-zasobnika/dane-spektro/
Powinieneś mieć teraz wiadro pełne ładnych spektrogramów!
Krok 4: Prześlij swój zbiór danych CSV
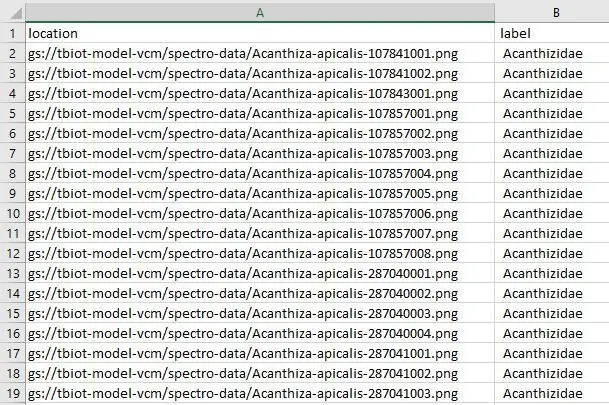
Teraz musimy przesłać plik model-labels.csv do katalogu „spectro-data/” w Google Storage, zasadniczo tak samo, jak w ostatnim kroku, przesyłasz tylko jeden plik zamiast wielu.
1. Kliknij nazwę swojego zasobnika na stronie Google Storage.
2. Wybierz przycisk PRZEŚLIJ PLIK i wybierz plik model-labels.csv, który utworzyłeś wcześniej.
Krok 5: Utwórz zbiór danych
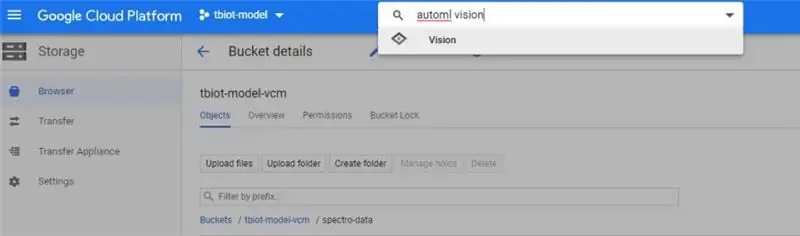
1. Najpierw musisz znaleźć API AutoML VIsion, może to być trochę trudne! Najprostszym sposobem jest wyszukanie „automl vision” na pasku wyszukiwania w pamięci Google Cloud (na zdjęciu).
2. Po kliknięciu łącza API będziesz musiał włączyć API.
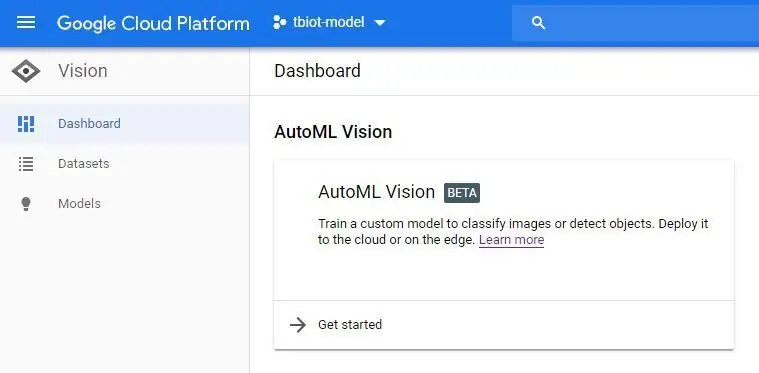
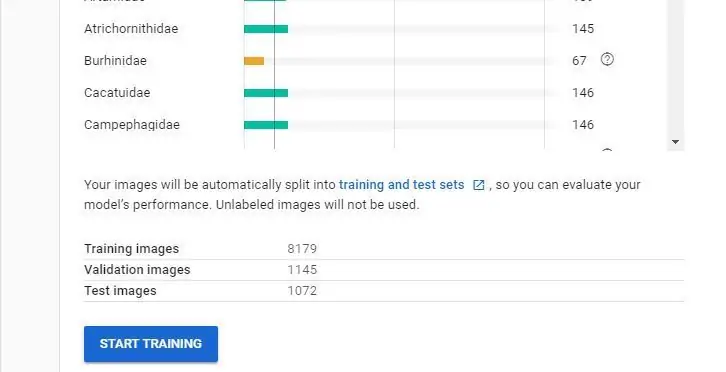
3. Teraz będziesz w panelu AutoML Vision (na zdjęciu) kliknij przycisk nowego zestawu danych i wybierz Pojedyncza etykieta i opcję „Wybierz plik CSV”. Następnie umieścisz link do pliku model-labels.csv w zasobniku na dane. Jeśli postępowałeś zgodnie z tym samouczkiem, będzie tak jak poniżej
gs://NazwaTwojegoZasobnika/dane-spektro/etykiety-modeliBal.csv
4. Następnie naciśnij przycisk Kontynuuj, aby utworzyć zestaw danych. Tworzenie może zająć trochę czasu.
Krok 6: Utwórz swój model AutoML
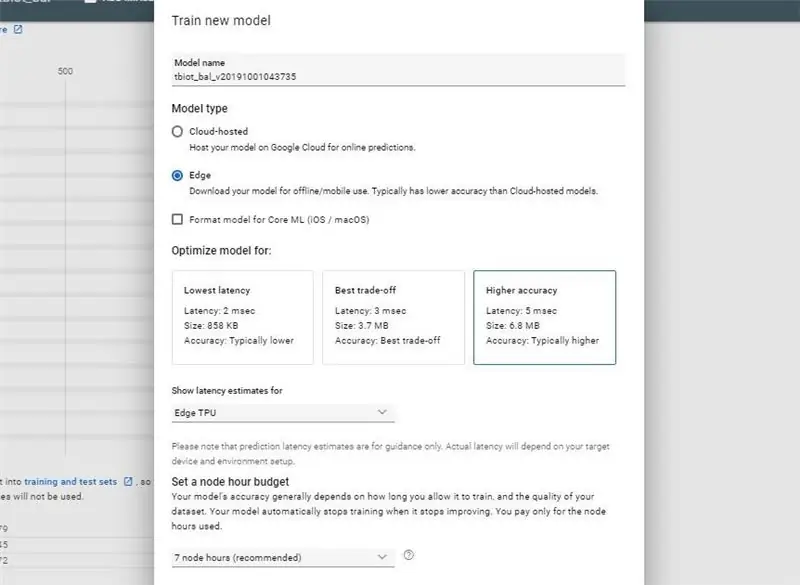

Po otrzymaniu wiadomości e-mail z informacją o utworzeniu zestawu danych możesz rozpocząć tworzenie nowego modelu.
- Naciśnij przycisk POCIĄG
- Wybierz typ modelu: Oszacowanie opóźnień krawędzi i modelu: Edge TPU i początkowo pozostaw inne opcje jako domyślne, ale możesz później poeksperymentować.
- Teraz Twój model będzie trenował, zajmie to trochę czasu i otrzymasz wiadomość e-mail, gdy będzie gotowy do pobrania.
Uwaga: jeśli przycisk trenowania jest niedostępny, możesz mieć problemy ze swoim zbiorem danych. Jeśli masz mniej niż 10 z każdej klasy (etykiety), system nie pozwoli Ci wyszkolić modela, więc być może będziesz musiał dodać dodatkowe obrazy. Jeśli potrzebujesz wyjaśnienia, warto zajrzeć do Google AutoML Video.
Krok 7: Przetestuj swój model
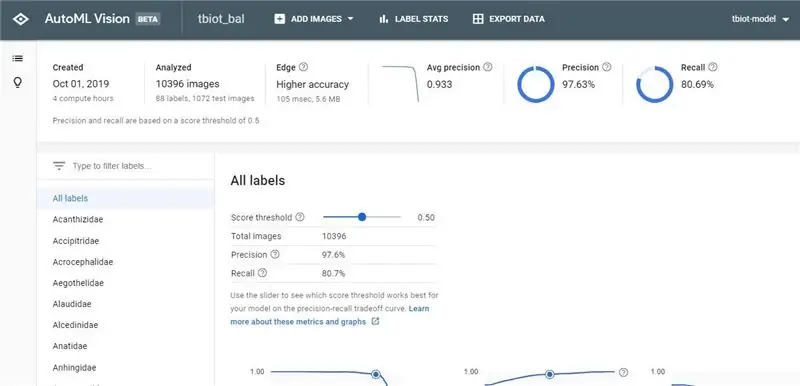
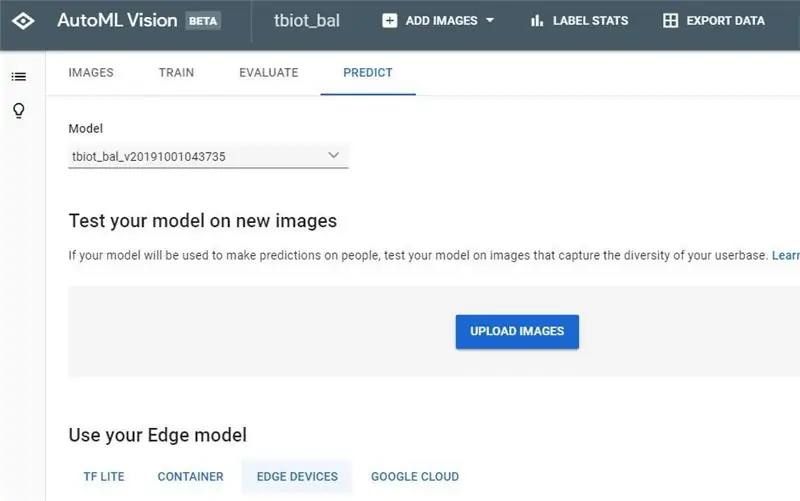
Po otrzymaniu wiadomości e-mail dotyczącej ukończenia modelu kliknij łącze, aby powrócić do interfejsu AutoML Vision API.
1. Teraz będziesz mógł zobaczyć swoje wyniki i macierz pomyłek dla swojego modelu.
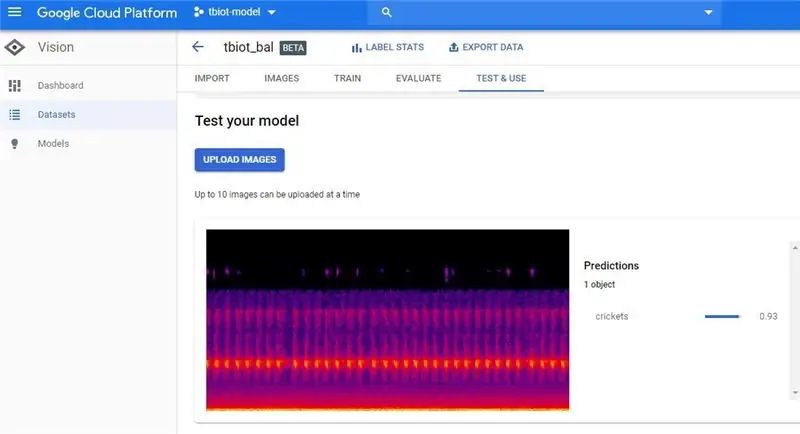
2. Następnym krokiem jest przetestowanie modelu, przejdź do „TESTUJ I UŻYJ” lub „PRZEWIDZAJ”. Dziwne wydaje się, że są tam 2 GUI użytkownika, oba zobrazowałem, ale obie opcje mają tę samą funkcjonalność.
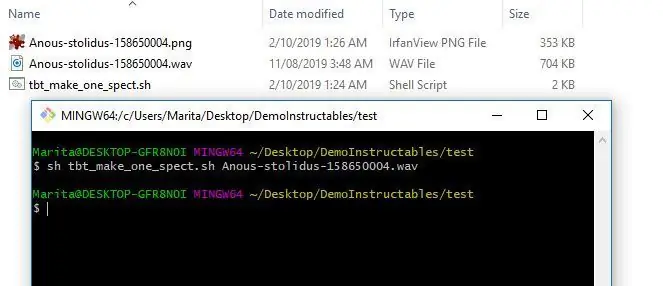
3. Teraz możesz przesłać testowy spektrogram. Aby wykonać pojedynczy spektrogram, możesz użyć programu tbt_make_one_spect.sh z ThinkBioT Github. Po prostu upuść go w folderze z wav, który chcesz przekonwertować na spektrogram, otwórz okno Git Bash (lub terminal) i użyj poniższego kodu, zastępując swoją nazwę pliku.
sh tbt_make_one_spect.sh twojaNazwaWav.wav
4. Teraz po prostu załaduj spektrogram i sprawdź swój wynik!
Krok 8: Zainstaluj swój model w ThinkBioT
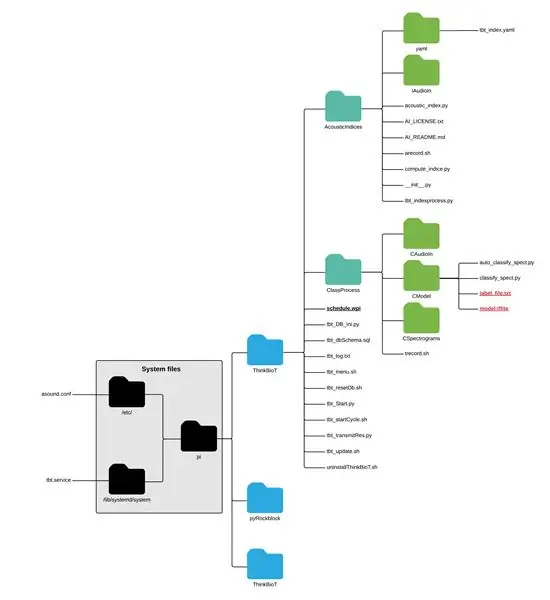
Aby użyć nowego błyszczącego modelu, po prostu upuść model i plik txt do folderu CModel;
pi > ThinkBioT > ClassProcess > CModel
Teraz jesteś gotowy do korzystania z ThinkBioT:)
**NB** Jeśli używasz modelu poza środowiskiem ThinkBioT, musisz edytować dokument etykiety, dodając liczby na początku każdego wiersza, ponieważ wbudowana funkcja „readlabels” najnowszych interpreterów tflite zakłada, że istnieją. Napisałem niestandardową funkcję w frameworku ThinkBioT classify_spect.py jako obejście, którą możesz wykorzystać we własnym kodzie:)
def ReadLabelFile(ścieżka_pliku):
licznik = 0 z open(ścieżka_pliku, 'r', kodowanie='utf-8') jako f: lines = f.readlines() ret = {} dla linii w liniach: ret[int(counter)] = line.strip () licznik = licznik + 1 powrót ret
Zalecana:
Kask ochronny Covid, część 1: wprowadzenie do obwodów Tinkercad!: 20 kroków (ze zdjęciami)

Kask ochronny Covid, część 1: wprowadzenie do obwodów Tinkercad!: Witaj, przyjacielu! W tej dwuczęściowej serii nauczymy się korzystać z obwodów Tinkercad - zabawnego, potężnego i edukacyjnego narzędzia do nauki działania obwodów! Jednym z najlepszych sposobów uczenia się jest robienie. Dlatego najpierw zaprojektujemy nasz własny projekt: th
Renowacja lamp nocnych Rayotron (część 2): 13 kroków

Renowacja nocnego światła Rayotron (część 2): Moja lampka nocna Rayotron została zainspirowana generatorem elektrostatycznym o napięciu pół miliona woltów, zaprojektowanym do wytwarzania wysokoenergetycznych promieni rentgenowskich do badań w dziedzinie fizyki atomowej. Oryginalny projekt wykorzystywał 12-woltowy zasilacz prądu stałego do zasilania małego elektronicznego jonizatora powietrza, który
Retro lampka nocna „Rayotron” (część 1): 16 kroków

Retro lampka nocna „Rayotron” (część 1): WprowadzenieW grudniu 1956 r. Atomic Laboratories reklamowało Rayotron jako „Pierwszy tani generator elektrostatyczny i akcelerator cząstek” dla nauczycieli przedmiotów ścisłych i hobbystów [1]. Rayotron był superwymiarowym, naładowanym gumowym paskiem
Część 1. Autonomiczny czujnik bioakustyczny ThinkBioT Budowa sprzętu: 13 kroków

Część 1. ThinkBioT Autonomous Bio-acoustic Sensor Hardware Build: ThinkBioT ma na celu zapewnienie ram programowo-sprzętowych, zaprojektowanych jako technologiczny szkielet wspierający dalsze badania, poprzez obsługę drobiazgów związanych z gromadzeniem, wstępnym przetwarzaniem, transmisją danych i zadaniami wizualizacji, badacz
Jak zbudować swoje pierwsze działanie dla Google Home (w 10 minut) Część 1: 10 kroków
Jak zbudować swoje pierwsze działanie dla Google Home (w 10 minut) Część 1: Cześć, To pierwszy z serii artykułów, które będę pisać, w których dowiemy się, jak tworzyć i wdrażać działania w Google. Właściwie to pracuję nad „działaniami w google” z ostatnich kilku miesięcy. Przejrzałem wiele artykułów dostępnych na
