Spisu treści:
- Krok 1: Warunki wstępne
- Krok 2: Warunki wstępne (nie)
- Krok 3: Warunki wstępne (nie)
- Krok 4: Warunki wstępne (nie)
- Krok 5: Oczyść Matlab, aby przygotować się do uruchomienia kodu
- Krok 6: Wybierz 10 normalnych obrazów oka i 10 obrazów z objawami retinopatii cukrzycowej
- Krok 7: Wybierz 10 normalnych obrazów oka i 10 obrazów z objawami retinopatii cukrzycowej (nie)
- Krok 8: Utwórz 2 zmienne (normalną i zdiagnozowaną) i ustaw je na 0
- Krok 9: Utwórz pętlę for, aby automatycznie przesyłać normalne obrazy
- Krok 10: Utwórz pętlę for, aby automatycznie przesyłać normalne obrazy (nie)
- Krok 11: Przytnij granice obrazu
- Krok 12: Utwórz obraz w skali szarości
- Krok 13: Utwórz skontrastowany obraz
- Krok 14: Popraw kontrast obrazu
- Krok 15: Utwórz średni filtr
- Krok 16: Połącz filtr uśredniający z skontrastowanym obrazem
- Krok 17: Utwórz nową maskę średniej, odejmując piksele
- Krok 18: Utwórz filtrowany obraz binarny
- Krok 19: Usuń mniejsze plamy znalezione w filtrowanych obrazach
- Krok 20: Utwórz element strukturyzujący dysk
- Krok 21: Wykonaj morfologiczne operacje zamknięcia
- Krok 22: Znajdź obiekty z łącznością co najmniej 8
- Krok 23: Znajdź maksymalną liczbę połączonych pikseli
- Krok 24: Ustaw maksymalne wartości pikseli na 0 i znajdź piksele z łącznością >= 26 pikseli
- Krok 25: Usuń naczynia krwionośne z obrazu
- Krok 26: Wyświetlanie figury
- Krok 27: Usuń naczynia i policz krople krwi
- Krok 28: Zdiagnozuj obraz siatkówki na podstawie liczby zidentyfikowanych skrzepów krwi
- Krok 29: Jeśli jest więcej niż 5 plamek…
- Krok 30: Powtórz proces filtrowania dla normalnych obrazów z wartościami cyfr obrazu 2 i 3
- Krok 31: Powtórz cały proces dla zdiagnozowanych obrazów
- Krok 32: Analiza statystyczna
- Krok 33: Znalezienie przedziału ufności
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:32.
- Ostatnio zmodyfikowany 2025-01-23 15:03.

(Patrz zarys kodu powyżej)
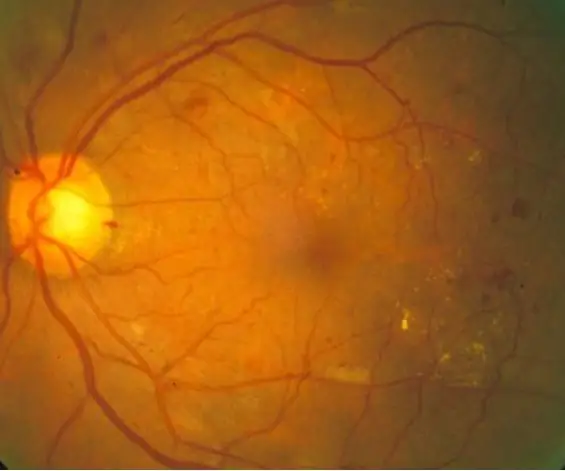
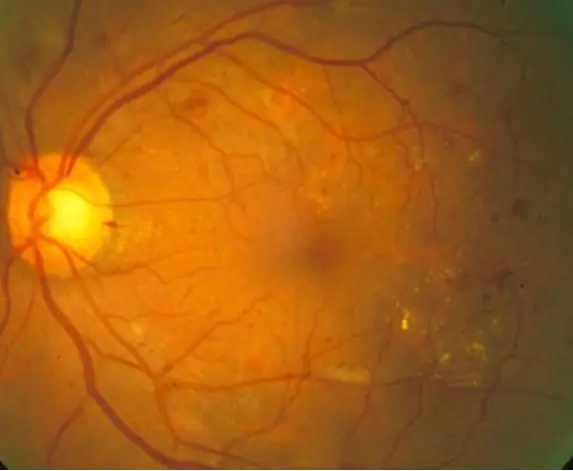
Retinopatia cukrzycowa to choroba oczu związana z cukrzycą, spowodowana wysokim poziomem cukru we krwi. Wysoki poziom cukru we krwi powoduje pęcznienie naczyń krwionośnych siatkówki, co prowadzi do powiększenia naczyń krwionośnych, a nawet nieszczelności naczyń, co prowadzi do ciemnych plam na obrazach siatkówki. Za pomocą tego kodu staramy się wykorzystać pojawianie się przecieków z naczyń krwionośnych jako wskaźnik podstawowej retinopatii cukrzycowej, chociaż w rzeczywistym świecie wymagane byłyby dalsze techniki diagnostyczne. Celem tego kodu jest automatyzacja przetwarzania obrazu i diagnozowania obrazów siatkówki w celu identyfikacji objawów retinopatii cukrzycowej widocznych w ciemnych plamach na obrazach siatkówki.
10 normalnych obrazów siatkówki i 10 zdiagnozowanych obrazów siatkówki zostało przetworzonych za pomocą kodu, który najpierw odczytuje i filtruje obrazy, a następnie określa ilościowo ciemne plamy w celu określenia, czy występują objawy retinopatii cukrzycowej, w oparciu o dany próg. Wyniki są następnie drukowane w oknie poleceń w celu interpretacji przez przeglądarkę.
Krok 1: Warunki wstępne
1. Upewnij się, że masz pobrany program MATLAB na swój komputer.
2. Pobierz plik txt znaleziony w linku. (Naciśnij „ctrl+s”, aby zapisać w tym samym katalogu, co kod MATLAB)
Krok 2: Warunki wstępne (nie)
4. Otwórz MATLAB i wpisz „uiimport” w oknie poleceń.
5. Wybierz plik Officialdiagnoses.txt i zaimportuj go do MATLAB jako macierz komórek.
6. Upewnij się, że widzisz „oficjalne diagnozy” jako zmienną w obszarze roboczym.
Krok 3: Warunki wstępne (nie)

7. Pobierz funkcję ModWald.m, którą można uzyskać z powyższego kodu lub pobrać go z Canvas.
(Kod dostarczony przez profesora Kinga i profesora Choi)
Krok 4: Warunki wstępne (nie)

8. Pobierz 400 surowych obrazów z sekcji danych projektu STARE.
Krok 5: Oczyść Matlab, aby przygotować się do uruchomienia kodu

Dodaj do kodu:
1. zamknij wszystko (Zamyka wszystkie wcześniej otwarte obrazy)
2. clearvars - z wyjątkiem oficjalnych diagnoz (Czyści wszystkie zmienne z wyjątkiem wcześniej zaimportowanego pliku txt oficjalnych diagnoz)
3. cclc (czyści okno poleceń)
Krok 6: Wybierz 10 normalnych obrazów oka i 10 obrazów z objawami retinopatii cukrzycowej
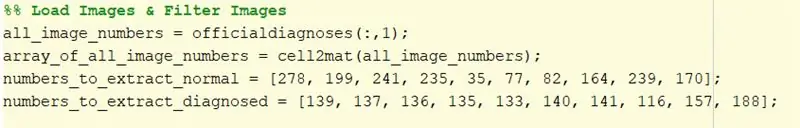
1. Weź plik tekstowy diagnostyki i wyodrębnij nazwy obrazów. Nazwy te znajdują się w pierwszej kolumnie pliku tekstowego, więc aby je wyodrębnić, wpisz „officialdiagnoses(:, 1)”. Macierz nazw obrazów została przypisana do zmiennej „wszystkie_liczby_obrazów”
2. Przekonwertuj zmienną all_image_numbers z tablicy komórek na tablicę macierzy za pomocą funkcji cell2mat
Krok 7: Wybierz 10 normalnych obrazów oka i 10 obrazów z objawami retinopatii cukrzycowej (nie)
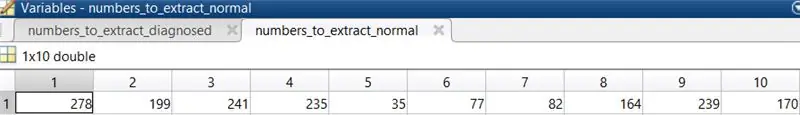
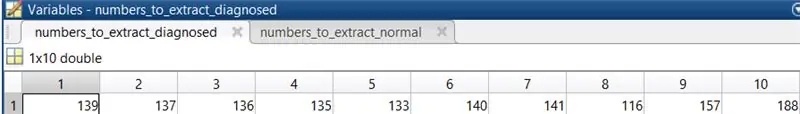
3. Wybierz 10 normalnych obrazów oczu, aby uruchomić kod. Wybrane w tym przypadku obrazy to 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Umieść te liczby w macierzy i przypisz je do zmiennej, która zostanie wywołana podczas ładowania obrazów.
4. Powtórz krok 3 dla obrazów siatkówki, u których zdiagnozowano retinopatię cukrzycową. Wybrane w tym przypadku obrazy to 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Krok 8: Utwórz 2 zmienne (normalną i zdiagnozowaną) i ustaw je na 0

Utwórz te zmienne przed pętlą for, aby zainicjować numery pętli.
Krok 9: Utwórz pętlę for, aby automatycznie przesyłać normalne obrazy

1. Utwórz pętlę for
2. Ustaw zmienną liczącą (w tym przypadku i) na macierz wartości 1-10. Ta zmienna zliczająca będzie używana do wywoływania każdego obrazu indywidualnie
3. Weź element i w macierzy obrazów, aby wyodrębnić i przekonwertować nazwę obrazu z ciągu na liczbę za pomocą funkcji num2str.
Znajdź liczbę cyfr występujących w nazwie obrazu za pomocą funkcji numel. Przypisz tę wartość do zmiennej cyfry_normalne. Liczba ta powinna wynosić 1 dla liczb jednocyfrowych, 2 dla liczb dwucyfrowych i 3 dla liczb trzycyfrowych. Te informacje będą używane do automatycznego wywoływania obrazów.
Krok 10: Utwórz pętlę for, aby automatycznie przesyłać normalne obrazy (nie)

3. Utwórz instrukcję if zawierającą wszystkie trzy możliwości z poprzednich kroków. Jeśli nazwa obrazu ma 1 cyfrę, obraz będzie nazwany „im000”, jeśli ma 2 cyfry, obraz zostanie nazwany „im00”, a jeśli będzie miał 3, obraz zostanie nazwany „im0”.
4. Pod każdą instrukcją if przypisz zmienną do imread „im” pod odpowiednią instrukcją if z odpowiednią liczbą zer (jak opisano powyżej), po której następuje i.
Krok 11: Przytnij granice obrazu
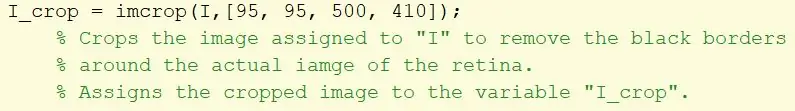
Weź oryginalny obraz i zastosuj filtr imcrop, aby wyeliminować czarne obramowania i przypisz do zmiennej I_crop. Prostokąt kadrowania określa się za pomocą macierzy [95, 95, 500, 410].
Krok 12: Utwórz obraz w skali szarości

Weź wykadrowany obraz i zastosuj filtr rbg2gray, aby zmienić obraz na skalę szarości. Przypisz ten obraz do zmiennej I2.
Krok 13: Utwórz skontrastowany obraz
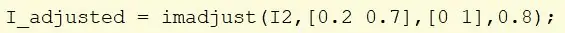
Zrób obraz I2 i użyj imadjust, aby przeskalować wartości intensywności.
Weź wartości mieszczące się w zakresie [0.2, 0.7] i przeskaluj je do [0, 1]. Gamma jest ustawiona na 0,8, aby obraz był jaśniejszy. Przypisz nowy obraz do I_adjusted.
Krok 14: Popraw kontrast obrazu
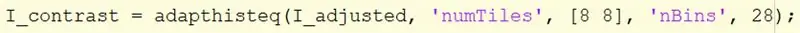
Zrób obraz I_adjusted i użyj funkcji adapthisteq, aby zwiększyć kontrast.
Składnia Adapthisteq wymaga nazwy obrazu I_adjusted, „numTiles”, rozmiaru numTiles, „nBins” i liczby pojemników. Rozmiar numTiles jest ustawiony na [8 8], dzieląc obraz na kafelki 8x8, a liczba pojemników jest ustawiona na 28. Przypisz obraz do I_constrast.
Krok 15: Utwórz średni filtr

Utwórz zmienną o nazwie 'meanfilt' za pomocą funkcji fspecial. Wprowadź „funkcję średniej”, aby utworzyć filtr uśredniający i wstaw [90 90] dla rozmiaru okna przesuwnego.
Krok 16: Połącz filtr uśredniający z skontrastowanym obrazem
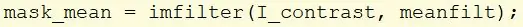
Utwórz nową zmienną o nazwie mask_mean i użyj funkcji imfilter, aby pobrać obraz I_contrast i zastosować wcześniej utworzony filtr średniej.
Krok 17: Utwórz nową maskę średniej, odejmując piksele

Utwórz zmienną o nazwie mask_mean2 i użyj funkcji imsubtract, aby odjąć wartość każdego piksela w I_contrast od odpowiedniego piksela w mask_mean.
Krok 18: Utwórz filtrowany obraz binarny

Zmień obrazy w skali szarości na czarno-białe za pomocą imbinarize. Input mask_mean2, ‘adaptive’, ‘ForegroundPolarity’, ‘dark’, ‘Sensitivity’, 0.6. Przypisz ten nowy obraz do mask_binarize.
Krok 19: Usuń mniejsze plamy znalezione w filtrowanych obrazach
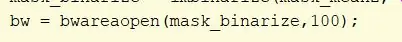
Usuń obiekty z łącznością mniejszą niż 100 pikseli za pomocą funkcji bwareaopen w mask_binarize i ustaw wartość progową na 100. Przypisz zmienną jako bw.
Krok 20: Utwórz element strukturyzujący dysk

Utwórz element strukturyzujący dysk (o promieniu 2) za pomocą funkcji strel. Przypisz go do se.
Krok 21: Wykonaj morfologiczne operacje zamknięcia

Weź bw i zastosuj funkcję imclose do elementu strukturalnego, aby wykonać morfologiczną operację zamknięcia na obiekcie.
Krok 22: Znajdź obiekty z łącznością co najmniej 8
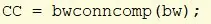
Weź bw i użyj bwconncomp, aby znaleźć obiekty z łącznością co najmniej 8 na obrazie. Przypisz numer wyjściowy do cc_1.
Krok 23: Znajdź maksymalną liczbę połączonych pikseli
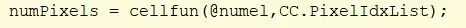
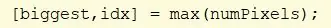
Użyj funkcji cellfun, aby wykonać funkcję „numel” na każdej komórce w CC. Znajduje to liczbę elementów w komórce PixelIdxList. Przypisz wartość do „numPixels”.
Znajdź maksymalne wartości w numPixels. Przypisz największe maksimum do „największego”, a indeks maksymalnej wartości do „idx”.
Krok 24: Ustaw maksymalne wartości pikseli na 0 i znajdź piksele z łącznością >= 26 pikseli
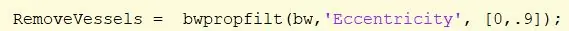



Ustaw piksele o największych wartościach w obrazie „bw” na 0, dzięki czemu piksele będą czarne.
Znajdź obiekty z łącznością co najmniej 26 pikseli na obrazie za pomocą bwconncomp. Przypisz do zmiennej cc_1.
Krok 25: Usuń naczynia krwionośne z obrazu
Usuń naczynia krwionośne nadal obecne na obrazie za pomocą funkcji bwpropfilt w zakresie [0, 0,9].
[0.9, 1] jest wykluczone, ponieważ wartości bliskie 1 wskazują linię. Przypisz do „Usuń naczynia”.
Krok 26: Wyświetlanie figury
Wyświetl każdy filtrowany obraz na wykresie podrzędnym. Pokaż. z danymi wejściowymi „border” i „tight” wyświetla każdy obraz w strukturze podrzędnej. Dodaj tytuł do każdego obrazu, aby odróżnić, który filtr został użyty.
Krok 27: Usuń naczynia i policz krople krwi
1. Weź „RemoveVessels” i zastosuj funkcję „Centroid” w rekwizytach regionu, aby zidentyfikować centroidy obiektów na obrazie. Obiekty te powinny odpowiadać skrzepom krwi obecnym na obrazie.
2. Policz liczbę zidentyfikowanych skrzepów krwi, biorąc długość matrycy centroidu.
Krok 28: Zdiagnozuj obraz siatkówki na podstawie liczby zidentyfikowanych skrzepów krwi
Użyj instrukcji if, aby zdiagnozować obraz na podstawie liczby zidentyfikowanych skrzepów krwi.
Jeśli liczba zidentyfikowanych centroidów była mniejsza lub równa 5, obraz był identyfikowany jako normalny.
Jeśli liczba centroidów była większa niż 5, w obrazie rozpoznano retinopatię cukrzycową.
Wynik jest wypisywany w oknie poleceń za pomocą fprintf.
Krok 29: Jeśli jest więcej niż 5 plamek…
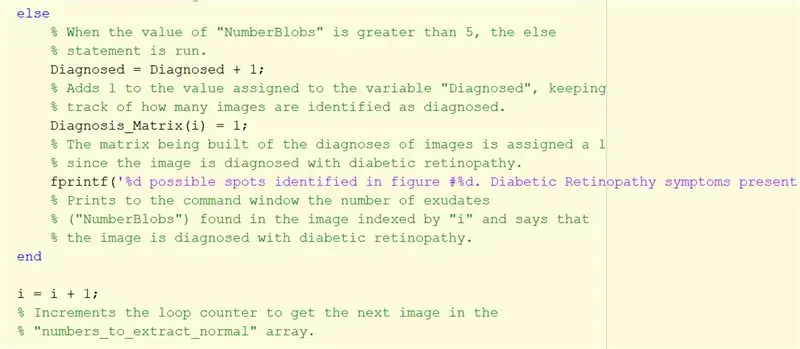
Powtórz powyższe instrukcje dla zdiagnozowanych obrazów jako inne stwierdzenie. Ta część zostanie uruchomiona, jeśli liczba obiektów blob jest większa niż 5.
Zakończ instrukcję if.
Krok 30: Powtórz proces filtrowania dla normalnych obrazów z wartościami cyfr obrazu 2 i 3
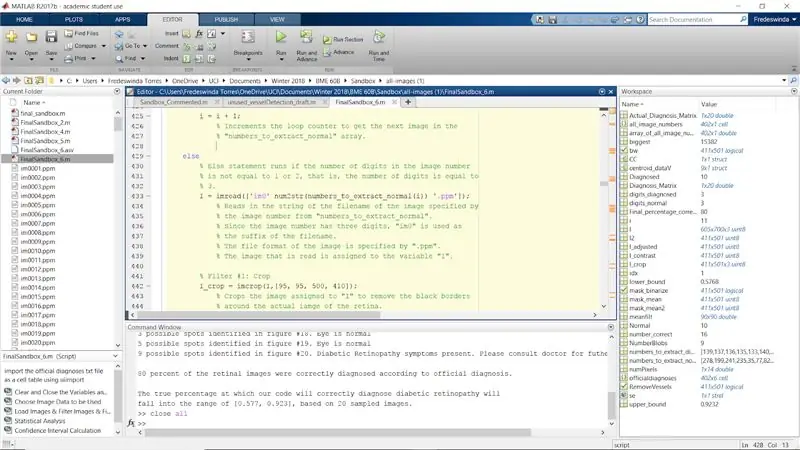
Powtórz ten proces dla pozostałej części oryginalnych instrukcji if, gdy liczba (liczba cyfr w numerze obrazu) jest równa 2 i 3. To kończy pętlę for dla normalnych obrazów.
Zakończ pętlę for.
Krok 31: Powtórz cały proces dla zdiagnozowanych obrazów
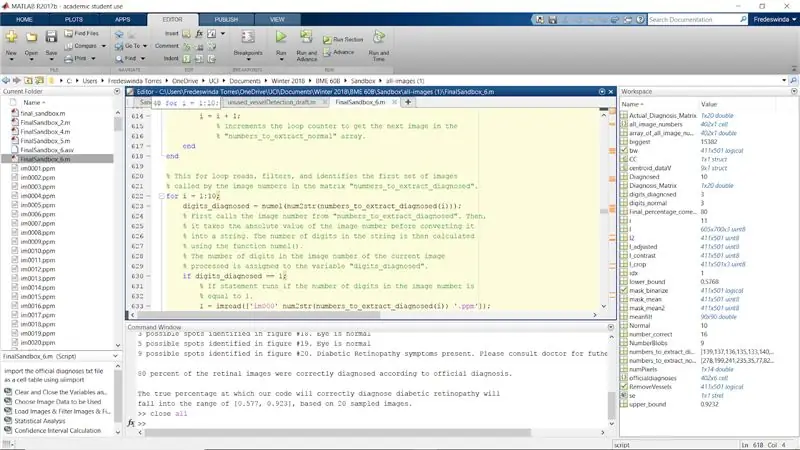
Powtórz cały proces, używając zdiagnozowanych obrazów wymienionych w macierzy „liczby_do_wydobycia_diagnozowane”.
Pamiętaj, aby przejrzeć każdą figurę (i) i zmienić ją na figurę (i+10), aby zdiagnozowane figury pojawiły się jako obrazy od 11 do 20.
Krok 32: Analiza statystyczna
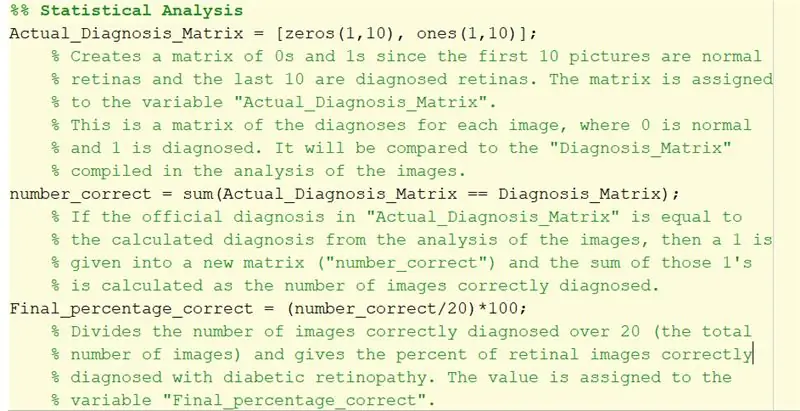
1. 'Actual_Diagnosis_Matrix' służy do porównania wyników z oficjalną diagnozą znalezioną w pliku txt. Pierwsze 10 zer wskazuje, że pierwsze 10 obrazów powinno być normalne. Ostatnie 10 obrazów wskazuje, że ostatnie 10 obrazów powinno być zaklasyfikowane jako retinopatia cukrzycowa.
2. Podwójny znak równości użyty do utworzenia „number_correct” tworzy logiczną tablicę, porównując wartość odpowiednich elementów „Actual_Diagnosis_Matrix” z „Diagnosis_Matrix” utworzonych z pętli for.
Dla każdego elementu pasującego do diagnozy zostanie dodane 1, co oznacza, że kod poprawnie zdiagnozował ten obraz. Jeśli jest niepoprawny, doda 0 do matrycy.
Następnie, biorąc sumę, która sumuje wszystkie jedynki. Innymi słowy, znajduje sumę prawidłowo zdiagnozowanych obrazów.
3. „Final_percentage_correct” to obliczony procent dokładności kodu zdiagnozowanej retinopatii cukrzycowej. Liczba prawidłowo zdiagnozowanych obrazów jest podzielona przez 20 (całkowita liczba obrazów) i pomnożona przez 100, aby obliczyć procent pomyślnych diagnoz.
Krok 33: Znalezienie przedziału ufności
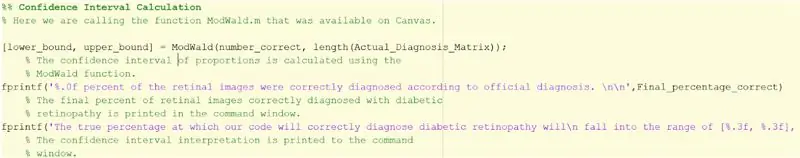
1. Upewnij się, że pobrałeś plik ModWald.m, aby wywołać go jako funkcję. Bez tej funkcji musiałbyś sam obliczyć przedział ufności za pomocą zmodyfikowanej metody Walda.
2. Funkcja ModWald posiada 2 wejścia, z których pierwsze to liczba poprawnie zidentyfikowanych obrazów, a drugie to łączna liczba obrazów.
3. Funkcja ModWald wyświetli dolną i górną granicę przedziału ufności proporcji dla dokładności próbkowanych danych. Innymi słowy, podasz przedział procentowy, w którym będzie leżał prawdziwy procent dokładności kodu.
4. Użyj fprintf poniżej, aby wyświetlić statystyki i przedział ufności w oknie poleceń.
> fprintf('%.0f procent obrazów siatkówki zostało prawidłowo zdiagnozowanych zgodnie z oficjalną diagnozą. \n\n', Final_percentage_correct)
> fprintf('Prawdziwy procent, przy którym nasz kod poprawnie zdiagnozuje retinopatię cukrzycową,\n mieści się w zakresie [%.3f, %.3f], na podstawie 20 pobranych próbek \n', lower_bound, upper_bound)
Zalecana:
Kontroluj swoją automatyczną bramę przesuwną za pomocą Home Assistant i ESPHome: 5 kroków (ze zdjęciami)

Kontroluj swoją automatyczną bramę przesuwną za pomocą Home Assistant i ESPHome: Poniższy artykuł zawiera informacje zwrotne na temat moich osobistych doświadczeń związanych z kontrolowaniem automatycznej bramy przesuwnej, którą zainstalowałem w moim domu. Brama ta, oznaczona marką „V2 Alfariss”, była wyposażona w kilka pilotów Phox V2 do sterowania. Też mam
Automatyczna żarówka za pomocą czujnika PIR: 3 kroki

Automatyczna żarówka z czujnikiem PIR: Cześć chłopaki! Tutaj przedstawiam automatyczne światło, które włącza się w zasięgu wzroku człowieka lub stworzenia. Wykorzystywany tutaj czujnik to wyjątkowo znany czujnik PIR. Jest to podstawowy obwód, który jest szybko dostępny w Internecie. Kupuję
Dyski twarde: diagnostyka, rozwiązywanie problemów i konserwacja: 3 kroki

Dyski twarde: diagnostyka, rozwiązywanie problemów i konserwacja: co to jest dysk twardy? - Mówiąc prościej, dysk twardy przechowuje wszystkie dane. Mieści dysk twardy, na którym fizycznie znajdują się wszystkie twoje pliki i foldery. Informacje są przechowywane na dysku magnetycznie, dzięki czemu pozostają na dysku nawet wtedy, gdy
Automatyczna zasłona/zasłona okienna za pomocą Arduino i LDR: 3 kroki

Automatyczna zasłona/zasłona okienna za pomocą Arduino i LDR: W tym samouczku zademonstrujemy, jak wykonać automatyczną roletę okienną za pomocą Arduino i modułu LDR. Za dnia zasłona/żaluzja zwinie się, a w nocy podwinie
Zapasy - Szybka diagnostyka komputera została zastąpiona, patrz poniżej: 6 kroków

Zapasy - Szybka diagnostyka komputera teraz zastąpiona, patrz poniżej: Jeśli kiedykolwiek będziesz musiał pracować na komputerze, którego nie masz przed sobą, potrzebujesz dokładnego spisu. Ta instrukcja pojawiła się wyłącznie dlatego, że kolega ma problem ze znalezieniem sterownika do komputera. Użyję Aida32 osobistego z http://majorgeeks.com
