Spisu treści:
- Krok 1: Korzystanie z Brain Box
- Krok 2: Wprowadzenie: Analiza przekrojów poprzecznych
- Krok 3: Konfiguracja funkcji: LevelCurveTracings.m
- Krok 4: Opracowanie algorytmu związanego: znajdowanie granic
- Krok 5: Opracowanie algorytmu związanego: generowanie zewnętrznej tablicy ograniczeń
- Krok 6: Opracowanie algorytmu wiązania: praca z punktem centralnym
- Krok 7: Opracowanie algorytmu związanego: gdy centroid nie jest wyśrodkowany
- Krok 8: Opracowanie algorytmu związanego: interferencja otworów
- Krok 9: Opracowanie algorytmu związanego: lokalizowanie otworów, finalizowanie granic mózgu i granic otworów
- Krok 10: Rejestrowanie danych: funkcja PatientFiles.m
- Krok 11: Logowanie danych do plików
- Krok 12: Rejestrowanie danych: Wyświetlanie wykresu objętości mózgu w czasie
- Krok 13: Zamykanie luk w poddziałkach: Subplotclose.m
- Krok 14: Główny kod: Czyszczenie wszystkiego i monitowanie o dane wejściowe
- Krok 15: Główny kod: wsadowe przetwarzanie obrazów
- Krok 16: Główny kod: dopełnienie
- Krok 17: Główny kod: określanie granic
- Krok 18: Kod Główny: Określanie Właściwego Wymiaru Z
- Krok 19: Główny kod: Określanie współrzędnych X i Y
- Krok 20: Kod główny: kreślenie struktury trójwymiarowej, znajdowanie objętości i rejestrowanie danych
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-31 10:23.
- Ostatnio zmodyfikowany 2025-01-23 15:03.
Zbliżanie się do granicy dłuższego ludzkiego życia spowodowało pojawienie się chorób, których cywilizacje nie widziały przed naszą. Wśród nich w 2017 roku na chorobę Alzheimera dotknęła około 5,3 miliona żyjących starszych Amerykanów lub około 1 na 10 starszych Amerykanów (https://www.alz.org/facts/) i niezliczonych innych z demencją. Aby pomóc w walce o zrozumienie, co dręczy naszych starszych, ten kod wyposaży przyszłych badaczy i ciekawskich w możliwość śledzenia objętości mózgu w czasie.
Krok 1: Korzystanie z Brain Box
Aby korzystać z pudełka mózgowego, wystarczy:
- Skany MRI mózgu oraz nazwa i format takich plików (powinny mieć mniej więcej takie same wymiary)
- Długość jednego skanu
- Odległość między każdą warstwą (skan MRI)
- Imię i nazwisko pacjenta (Podczas wprowadzania nie należy wprowadzać spacji i pisać imię i nazwisko z dużej litery, np.: ImięNazwisko)
Dzięki temu można śledzić indywidualne trendy w objętości mózgu w czasie. Dzięki temu oprogramowanie może śledzić dane dotyczące trendów choroby Alzheimera. Długość, którą zastosowaliśmy w próbie, wynosiła 180 mm dla długości jednego skanu i 5 mm dla odległości między skanami MRI, w oparciu o średnie wartości.
Jednak zastosowanie pudełka mózgowego nie musi ograniczać się do tego jednego zadania. Jeśli przekroje poprzeczne danej bryły są zdjęciami, jak sam guz, trendy zmian objętości dla nich można również śledzić w oprogramowaniu.
Krok 2: Wprowadzenie: Analiza przekrojów poprzecznych
W strukturach trójwymiarowych dwuwymiarowe płaszczyzny, z których się składają, nazwano by przekrojami poprzecznymi. Wyobraź sobie, że stos kartek tworzy prostokątny graniastosłup, wtedy każdy kawałek papieru byłby przekrojem papieru. W wyobrażaniu sobie mózgu stosujemy ten sam tok myślenia. MRI (obrazowanie metodą rezonansu magnetycznego) (patrz informacje na temat MRI) rejestruje przekroje mózgu, a korzystając z granic określonych w każdej „warstwie” mózgu, możemy zbudować strukturę do modelowania i określania objętości mózgu. Musimy jednak najpierw zbudować funkcję, która dostarczy informacji o takich granicach.
Krok 3: Konfiguracja funkcji: LevelCurveTracings.m
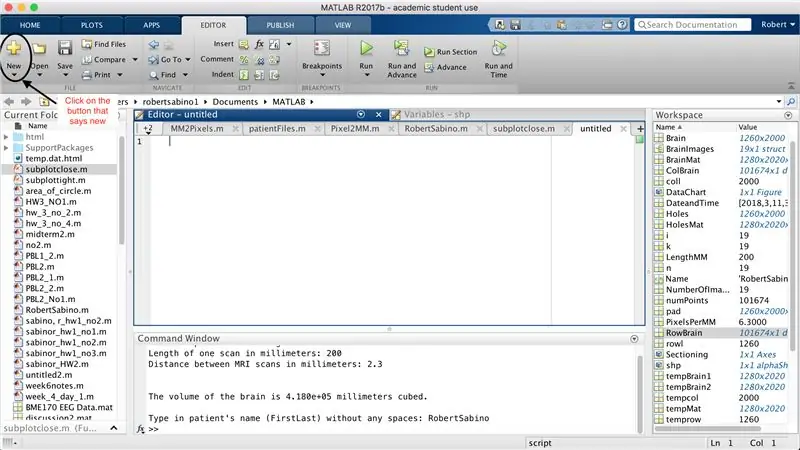
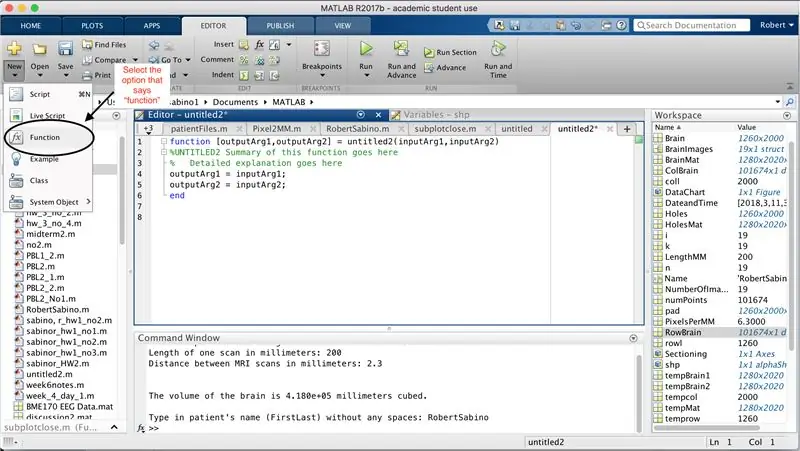
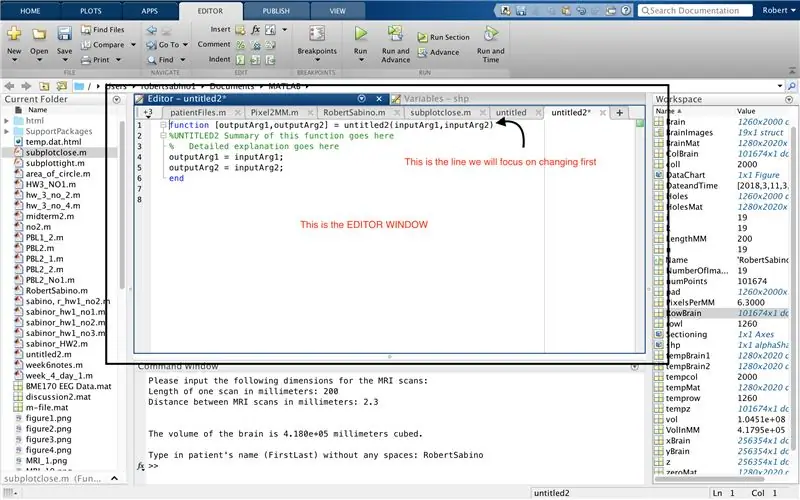

Najpierw upewnij się, że twój komputer ma pobrany MATLAB_R2017b (pobierz tutaj) i otwórz MATLAB. W interfejsie MATLAB kliknij przycisk w lewym górnym rogu okna z napisem „Nowy” z pogrubionym żółtym znakiem plus i wybierz opcję „funkcja”, aby otworzyć w oknie edytora spację podobną do tej w trzecie zdjęcie. Skoncentrujemy się na zmianie pierwszej linii, aby skonfigurować funkcję. Tam, gdzie jest napisane "outputArg1", zamień go na "mózg", "outputArg2", aby powiedzieć "dziury", "untitled2" na "exp2" i "inputArg1" na "obraz" i usuń "inputArg2". Masz teraz funkcję, którą można wywołać za pomocą "exp2", przyjmując jeden argument "image" i wyprowadzając granice "mózgu" i "dziur". Pierwsza linia funkcji powinna przypominać linię z czwartego rysunku. Usuń cały kod poniżej tej początkowej linii.
Krok 4: Opracowanie algorytmu związanego: znajdowanie granic

Wpisz kod w następujący sposób poniżej wiersza. Ta sekcja funkcji wykonuje następujące wiersze po wierszu.
- Załaduj obraz "image" do zmiennej "mri".
- Przekształć „mri” w obraz złożony z wartości z zakresu liczb do jedynek i zer (inaczej Binaryzacja) w oparciu o ustaloną wartość progową. Jeśli wartość w pikselu jest równa lub większa niż 0,1, jest ustawiana na jeden, jeśli nie, wartość w tym pikselu jest ustawiana na zero.
- Kolejne cztery wiersze zamieniają 10 kolumn i wierszy na krawędziach warstwy skanowania MRI w zera, aby uniknąć odczytywania niewłaściwych wartości jako tworzenia granic (jak nauczyliśmy się podczas eksperymentowania z kodem).
- W ostatnim wierszu bwboundaries śledzi granice zbinaryzowanego obrazu „mri” i ustawia go na „b”, tablicę z elementami, których indeksy odpowiadają tym z granic ustawionych na jeden.
Krok 5: Opracowanie algorytmu związanego: generowanie zewnętrznej tablicy ograniczeń
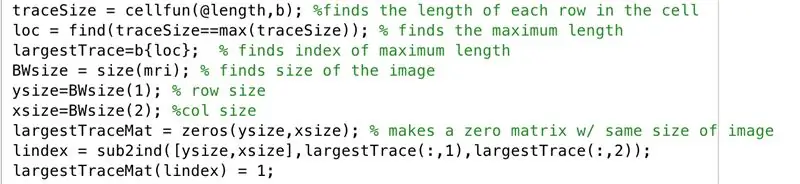
Śledź dalej w oknie edytora z następującym kodem na obrazku. Ta sekcja kodu wykonuje następujące wiersze.
- Znajdź długość każdego z wierszy zbinaryzowanego obrazu „b” (cellfun stosuje długość funkcji do każdego wiersza).
- Ustaw „loc”, aby zapisać maksymalne długości.
- Znajdź indeks maksymalnej długości, ustawiony do przechowywania w „largestTrace”.
- Znajdź rozmiar obrazu „mri”, który składa się z tego samego rozmiaru co „b” i ustaw na „BWsize”.
- Znajdź liczbę wierszy w tablicy obrazu, ustaw na „ysize”.
- Znajdź liczbę kolumn w tablicy obrazu, ustaw na „xsize”.
- Wygeneruj tablicę „largestTraceMat”, macierz zer „ysize” na „xsize”.
- Znajdź równoważny indeks z wartości indeksowanych odpowiadających tym, gdzie znajdowały się największe wartości Trace x i y, przechowuj w wektorze „lindex”.
- W macierzy zer „largestTraceMat” zamienia elementy w indeksach, które odpowiadają wartościom indeksu przechowywanym jako elementy w „lindex” w jedynki.
Tak więc tablica logiczna "largestTraceMat" ma największy ograniczony obszar danego przekroju skanu mózgu wykreślony jako jedynki z tłem zerowym
Krok 6: Opracowanie algorytmu wiązania: praca z punktem centralnym

Następnie musimy sprawdzić, czy przekrój składa się z więcej niż jednego regionu (największego). Testując wyrównanie centroidu największego regionu, możemy zobaczyć, czy istnieje jeden ciągły region, który dawałby bardziej wyśrodkowany centroid lub możliwość wielu regionów.
- Użyj "regionProps", aby znaleźć informacje o obecnych centroidach, ustaw równą tablicy struktury "tempStruct"
- Utwórz tablicę „centroids” z danymi z pola „centroid” połączonymi pionowo
- Weź wartości z drugiej kolumny „centroids” (współrzędne wymiaru poziomego)
- Uruchom filtr, aby sprawdzić wyrównanie środka ciężkości do środka w poziomie
Krok 7: Opracowanie algorytmu związanego: gdy centroid nie jest wyśrodkowany
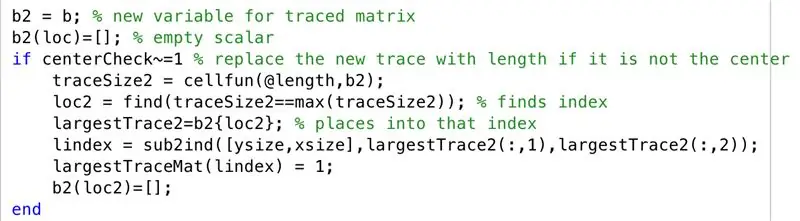
W scenariuszu, w którym centroid największego regionu śledzenia nie jest wyśrodkowany, wykonujemy następujące kroki. Jak zaobserwowaliśmy na skanach MRI, tendencję do przedstawiania półkul mózgu w przekroju poprzecznym, gdy nie są one przyległe, więc teraz kontynuujemy wykreślanie drugiego największego śladu wraz z największym śladem w „largestTraceMat”
- Ustaw macierz śledzoną na nową zmienną "b2"
- Zainicjuj pustą macierz „b2”, z zestawem indeksowanym przez „loc”
- Utwórz warunek, gdy centroid nie jest wyśrodkowany (tj. warstwa wielu regionów)
- Ustaw nowy rozmiar śledzenia do znalezienia dla każdego wiersza (traceSize2)
- Ustaw „loc2”, aby znaleźć indeksy, w których występują granice
- Niech komórki określone przez „loc2” w „b2” będą równe „largestTrace2”
- Konwertuj indeksy na indeksy, ustaw na „lindex”
- Zmień elementy odpowiadające „lindex” w „largestTraceMat” na 1
- Zainicjuj pustą macierz „b2”, z zestawem indeksowanym przez „loc2”
Krok 8: Opracowanie algorytmu związanego: interferencja otworów

W radzeniu sobie z dziurami, wartości przechowywane w „b2” śledziły struktury inne niż największy ślad, a wykreślenie ich na wypełnionym formularzu „largestTraceMat” ujawni, gdzie znajdują się dziury w obszarach mózgu.
- Utwórz tablicę „filledMat”, która jest wypełnioną formą „largestTraceMat”
- Utwórz tablicę "interferenceMat", tablicę zer "ysize" przez "xsize"
- Utwórz tablicę „interferenceloc”, aby przechowywać wartości z „b2” połączone pionowo
- Utwórz tablicę „lindex”, aby przechowywać indeksy odpowiadające „interferenceloc”
- Dla indeksów w "interferenceMat", które odpowiadają "lindex", ustaw wartość na 1, tworząc inny ograniczony region
Krok 9: Opracowanie algorytmu związanego: lokalizowanie otworów, finalizowanie granic mózgu i granic otworów

- Ustaw tablicę "tempMat" równą "interferenceMat" plus "filledMat", w ten sposób dodając do siebie każdą wartość w macierzy
- Ustaw tablicę "holesLoc" równą indeksom, gdzie "interferenceMat" i "filledMat" były równe jeden
- Ustaw "holesMat" jako zerową macierz wymiarów "ysize" x "xsize"
- Ustaw indeksy w "holesMat", które są równe "holesLoc" jako jedynki
- Ustaw "mózg" na "largestTraceMat"
- Ustaw "holes" na "holesMat"
Dzięki ustaleniu, w którym wartości dodanych macierzy były równe 2, lokalizacje otworów zostały łatwo zabezpieczone i naniesione na pustą macierz.
Krok 10: Rejestrowanie danych: funkcja PatientFiles.m
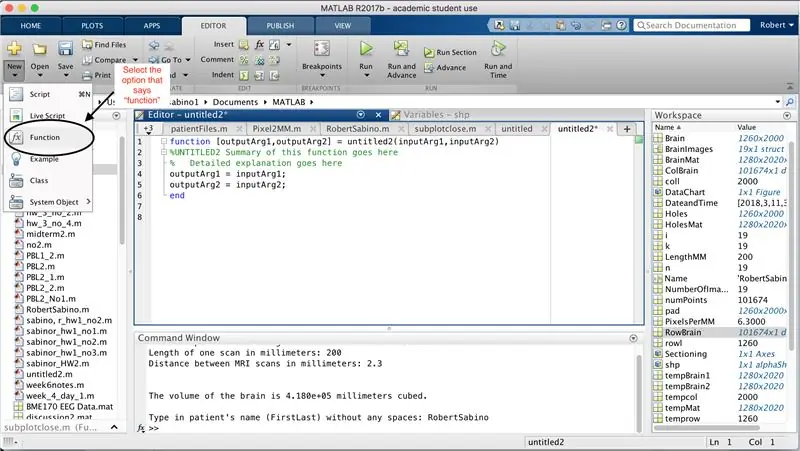
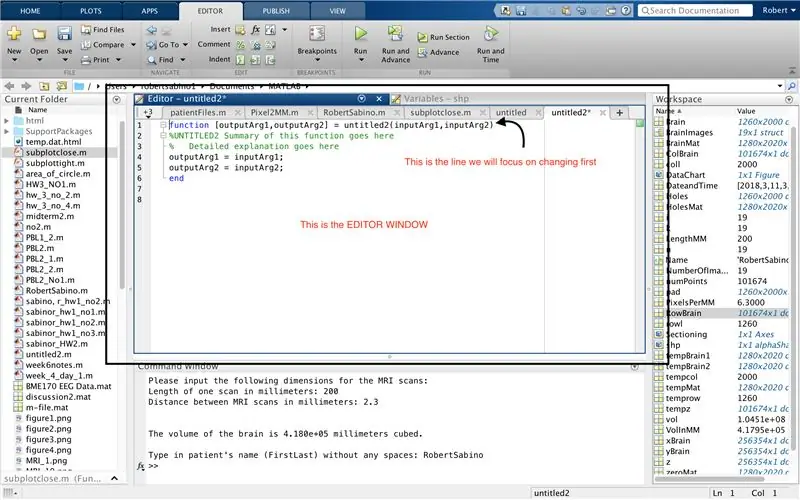

Podobnie jak w przypadku konfiguracji ostatniej funkcji, kliknij przycisk w lewym górnym rogu okna z napisem „Nowy” z pogrubionym żółtym znakiem plus i wybierz opcję „funkcja”, aby otworzyć w oknie edytora przestrzeń, która przypomina to na trzecim zdjęciu. W pierwszym wierszu usuń macierz wyjściową i zastąp tylko "wyjście", zamień "bez tytułu2" na "Pliki pacjenta", usuń wszystkie argumenty wejściowe i zamiast tego postępuj zgodnie z formatowaniem określonym na czwartym obrazie wiersza kodu. Pierwszy wiersz tej funkcji powinien pasować do formatowania obrazu.
Krok 11: Logowanie danych do plików
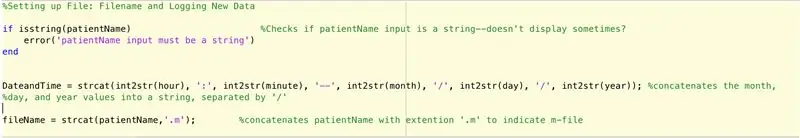

Aby skonfigurować plik do rejestrowania danych znalezionych przez funkcję main (jeszcze do opisania), musimy wykonać następujące kroki (zgodnie z kodem linia po linii).
- Sprawdź, czy dane wejściowe dla nazwa_pacjenta są ciągiem.
- Jeśli nie jest to ciąg, wyświetl, że dane wejściowe nazwa_pacjenta powinny być ciągiem.
- Zakończ instrukcję if (zapobiegaj błędom).
- Skonfiguruj ciąg znaków „DateandTime”, który da następujący format: godzina:minuty - miesiąc/dzień/rok.
- Ustaw zmienną fileName na następującą: nazwa_pacjenta.m.
Przejdźmy teraz do następnej sekcji funkcji: Czy plik o tej nazwie już istnieje?
1) Załóżmy, że plik o tej nazwie już istnieje:
- Uruchom plik, aby pobrać wartości z przeszłości w kolejce
- Dodaj dane "DateandTime" bieżącej iteracji jako nową komórkę w tablicy komórek o wartościach x (indeks end+1)
- Dodaj bieżącą wartość „brainVolume” jako nową komórkę w tablicy komórek wartości y (index end+1)
- Zapisz aktualne zmienne załadowane do pliku.
2) Załóżmy, że plik o tej nazwie nie istnieje:
- Utwórz nowy plik o nazwie zapisanej w zmiennej "patientName"
- Dodaj bieżące dane „Data i godzina” jako komórkę do pustej tablicy komórek zawierającej wartości x
- Dodaj bieżące dane „brainVolume” jako komórkę do pustej tablicy komórek z wartościami y
- Zapisz aktualne zmienne załadowane do pliku.
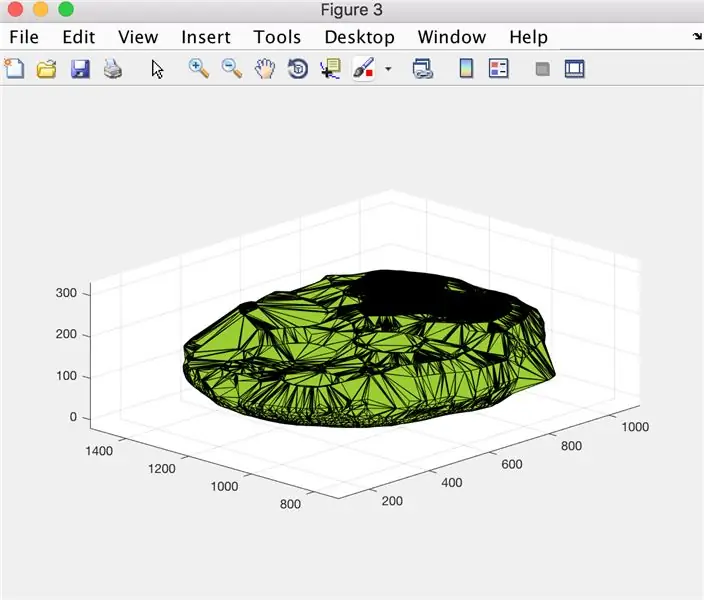
Krok 12: Rejestrowanie danych: Wyświetlanie wykresu objętości mózgu w czasie

- Przekształć tablicę wartości x (xVals) w tablicę kategorii (xValsCategorical), aby umożliwić kreślenie
- Okno generowania figury 5
- Wykreśl punkty wyznaczone przez „xValsCategorical” i „yVals” (zawierające objętość mózgu), używając pustych kółek do wskazania punktów i połączonych liniami przerywanymi
- Zatytułuj wykres jako: nazwa_pacjenta Dane dotyczące objętości mózgu
- Oznacz oś x, jak pokazano na rysunku!
- Oznacz oś y, jak pokazano na rysunku
- Niech liczba 5 równa się produkcji
Na tej podstawie wywoływana funkcja nazwa_pacjenta da plik z edytowanymi danymi śledzącymi objętość mózgu w czasie oraz wykres przedstawiający trendy.
Krok 13: Zamykanie luk w poddziałkach: Subplotclose.m

Funkcja, zaadaptowana z kodu ze strony https://www.briandalessandro.com, służy do zamykania luk pomiędzy figurami podrzędnymi w głównym kodzie, gdy tworzone są figury wyświetlające obrazy MRI i warstwy mózgu. Funkcja subplot używana w subplotclose.m dostosowuje pozycję danych wykresów podrzędnych, aby ściśle przylegały do siebie w aspekcie dłuższego wymiaru. Na przykład, jeśli kod przewiduje macierz 7 x 3, wiersze będą pasować do siebie, ponieważ wymiar wiersza jest dłuższy. Jeśli kod przewiduje macierz 3 x 7, kolumny będą pasować ściśle, z przerwami w wierszach, jak pokazano na rysunkach naszego głównego kodu.
Krok 14: Główny kod: Czyszczenie wszystkiego i monitowanie o dane wejściowe

Aby uruchomić główny kod, kliknij ten sam przycisk, który mówi „Nowy” w lewym górnym rogu okna i wybierz „Skrypt” zamiast „Funkcja” z poprzednich sekcji. Wpisz kod, jak pokazano na obrazku w oknie edytora. Linie kodu wykonują następujące zadania w kolejności:
- Zamknij wszystkie otwarte pliki z wyjątkiem 0, 1 i 2.
- Zamknij wszystkie okna z figurami.
- Wyczyść wszystkie zmienne w obszarze roboczym.
- Wyczyść okno poleceń.
- Wyświetl w oknie poleceń: Wprowadź następujące wymiary dla skanów MRI:
- W nowym wierszu w oknie poleceń zapytaj: Długość jednego skanu w milimetrach:. Odpowiedź wprowadzona przez użytkownika zostanie ustawiona na zmienną „lengthMM”.
- W nowej linii zapytaj: Odległość między skanami MRI w milimetrach:. Odpowiedź wprowadzona przez użytkownika zostanie ustawiona na zmienną „ZStacks”.
Krok 15: Główny kod: wsadowe przetwarzanie obrazów

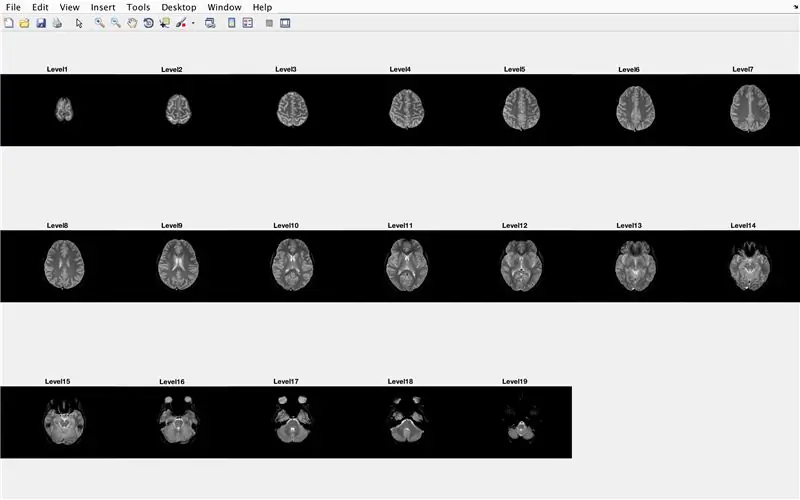
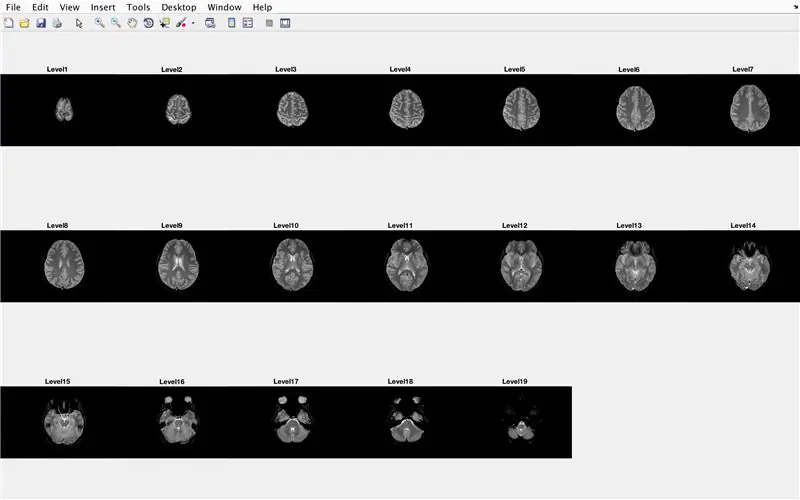
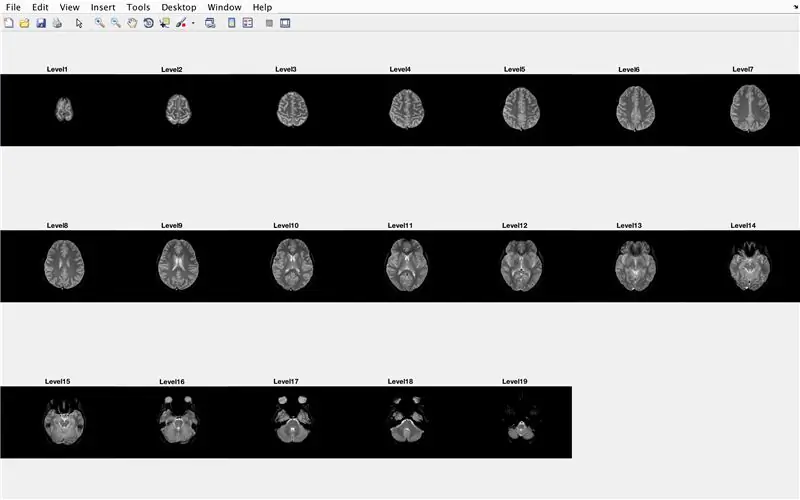
W tej sekcji kod załaduje obrazy (składające się ze skanów MRI przekrojów poprzecznych mózgu) i zapisze nazwy każdego pliku obrazu w zmiennej „Baza” i wyświetli każdy ze skanów MRI. Postępuj zgodnie z kodem na zdjęciu, który wykonuje następujące czynności:
- Utwórz tablicę struktury „BrainImages”, która zawiera informacje o wszystkich plikach w bieżącym folderze, które pasują do formatu nazwy MRI_().png
- Ustaw zmienną "NumberofImages" równą liczbie elementów w tablicy struktury "BrainImages"
- Otwórz okno figury 1
- Ustaw pętlę for, aby przejść przez liczbę obrazów zliczonych w pliku
- Dla każdej pętli "CurrentImage" jest odpowiednią nazwą każdego pliku MRI_i.png, z numerem iteracji jako 'i'
- Wygeneruj wykres podrzędny 3 x 7, aby wyświetlić 19 obrazów do załadowania przez „imshow”
- Wyświetl każdy obraz jako kolejny element w oknie figury podrzędnej
- Zatytułuj każdy element podrzędny jako Level_, gdzie puste to numer iteracji pętli for.
- Zakończ pętlę for (unikając błędu)
Spowoduje to wyświetlenie w oknie rysunku 1 wszystkich skanów MRI w postaci surowej w konfiguracji 3 x 7 bez przerw w orientacji x.
Krok 16: Główny kod: dopełnienie

Dzięki dopełnianiu unikamy problemu drobnych rozbieżności w rozmiarach obrazu, które mogą powodować błąd niezgodności wymiarów w przypadku, gdy jeden obraz jest nieco większy od drugiego.
- Otwórz okno figury 2
- Załaduj macierz obrazu z MRI_1-p.webp" />
- Znajdź rozmiar macierzy obrazu i ustaw „OriginalXPixels” (dla liczby wierszy) i „OriginalYPixels” (dla liczby kolumn)
- Ustaw macierz "BrainMat" tak, aby składała się ze wszystkich zer z 20 dodatkowymi wierszami i 20 dodatkowymi kolumnami dla każdej płaszczyzny oraz 19 całkowitymi przekrojami poprzecznymi, po jednym na płaszczyznę.
- Skonfiguruj "HolesMat", aby składał się z tej samej trójwymiarowej tablicy zer, aby później wprowadzić współrzędne otworu
- Utwórz "zeroMat" o rozmiarze pola plus dwadzieścia wierszy i dwadzieścia kolumn, dwuwymiarową tablicę zer.
Krok 17: Główny kod: określanie granic
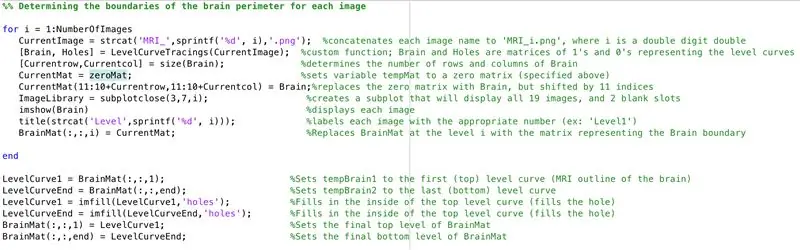
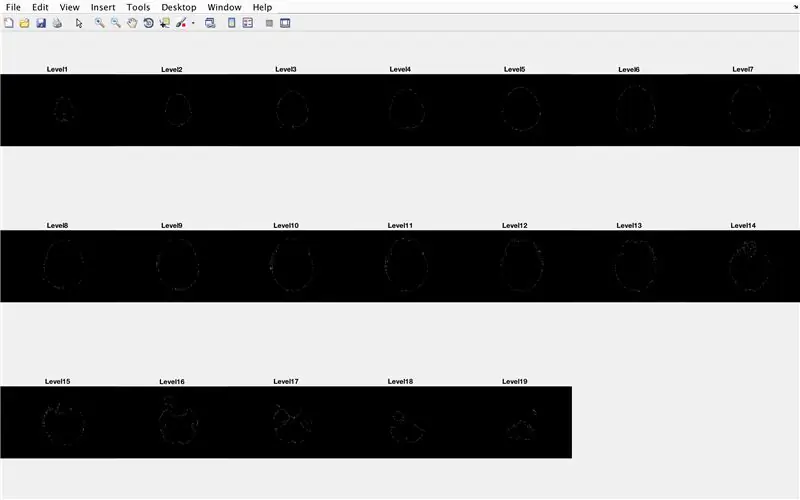
- Ustaw pętlę for, aby przejść przez dane z każdego załadowanego wcześniej obrazu
- W ten sam sposób przetwarzania wsadowego wcześniej, "CurrentImage" ładuje pliki z "MRI_i.png", gdzie i jest numerem iteracji
- Uruchom każdy obraz za pomocą utworzonej wcześniej funkcji przetwarzania „LevelCurveTracings2.m”
- Znajdź rozmiar wyjścia „Mózg”, ustaw liczbę wierszy na „Currentrow” i liczbę kolumn na „Currentcolumns”
- Ustaw "CurrentMat" na macierz zer o wymiarach określonych przez "Currentrow" i "Currentcolumns"
- Wyśrodkuj dane z „Mózgu” w „CurrentMat”, z marginesem 10 rzędów ze wszystkich stron
- Wygeneruj wykres pomocniczy o wymiarach 3 x 7, aby wyświetlić granice obrazów
- Nazwij każdy z elementów podrzędnych w oknie rysunku
- Wygeneruj trójwymiarową macierz „BrainMat” składającą się z każdej warstwy granicznej „CurrentMat”
- Zakończ pętlę for (aby uniknąć błędów)
Podrozdział następujący po wypełnieniu otworów pozostawionych na górze i na dole proponowanego trójwymiarowego kształtu
- Ustaw "LevelCurve1" równą pierwszej warstwie "BrainMat" (spód bryły)
- Ustaw "LevelCurveEnd" równą ostatniej warstwie "BrainMat" (góra bryły)
- Zastąp „LevelCurve1” wypełnioną warstwą
- Zastąp "LevelCurveEnd" wypełnioną warstwą
- Ustaw wypełnioną warstwę jako dolną warstwę "BrainMat"
- Ustaw wypełnioną warstwę jako górną warstwę „BrainMat”
Krok 18: Kod Główny: Określanie Właściwego Wymiaru Z

Pierwsze trzy wiersze polegają na ustawieniu pustej tablicy "z" i wykonaniu prostych operacji konwersji (podziel piksele według długości), aby uzyskać prawidłowy odczyt objętości w mm^3
- Utwórz pętlę for, aby przechodzić przez każdą warstwę
- Znajdź liczbę jedynek w danej warstwie
- Przekształć współrzędne z dla tych na wartości, które są przeskalowane we właściwym stosunku, ustaw na „tempz”, wektor kolumnowy
- Dodaj wartość z dla krzywej poziomu do wektora z
Dzięki temu współrzędne z są odpowiednio dopasowane.
Krok 19: Główny kod: Określanie współrzędnych X i Y

Teraz wyznacz pozycje xiy każdego z punktów na granicach.
- Zainicjuj „xBrain” jako pustą tablicę
- Zainicjuj "yBrain" jako pustą tablicę
- Skonfiguruj pętlę for, aby przejść przez każdy załadowany obraz
- Skompiluj macierz dwukolumnową, aby przechowywać współrzędne planarne każdego punktu w ograniczeniu, reprezentowane przez wektory kolumnowe „RowBrain” i „ColumnBrain”
- Dołącz "xBrain" do aktualnie znalezionych współrzędnych "RowBrain"
- Dołącz "yBrain" z aktualnie znalezionymi współrzędnymi "ColumnBrain"
- Zakończ pętlę for (aby uniknąć błędu)
Krok 20: Kod główny: kreślenie struktury trójwymiarowej, znajdowanie objętości i rejestrowanie danych
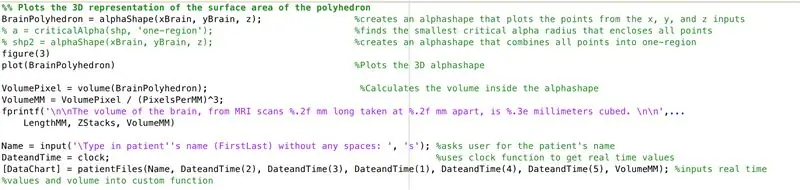

Korzystając z funkcji alphaShape stworzymy trójwymiarową strukturę, z której będziemy mogli obliczyć objętość mózgu.
- Użyj funkcji alphaShape, podłącz wektory "xBrain", "yBrain" i "z" dla współrzędnych x, y i z i ustaw równą "BrainPolyhedron"
- Otwórz okno figury 3
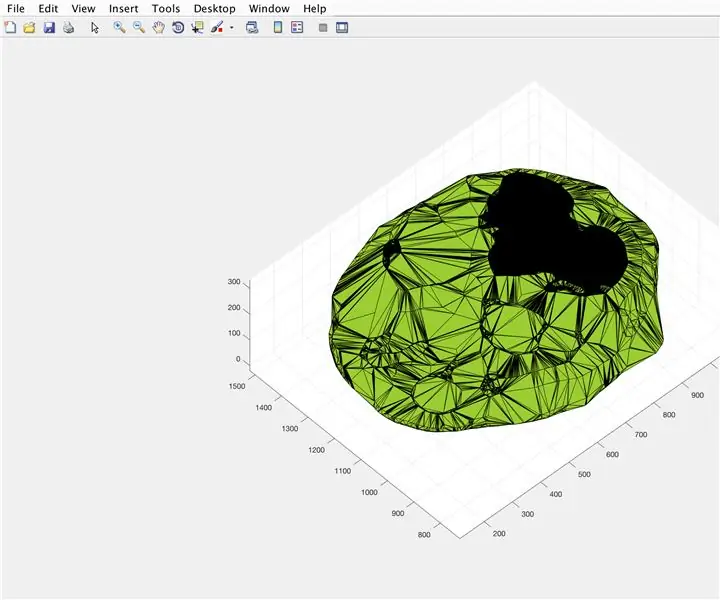
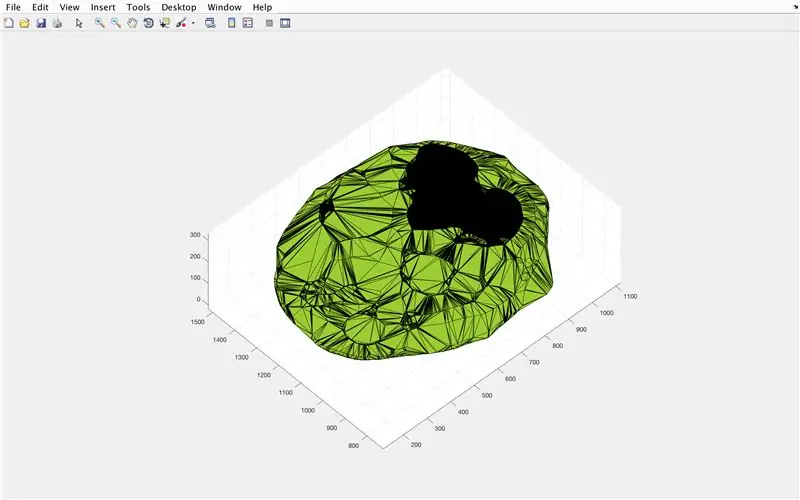
- Narysuj obliczony kształt alfa „Mózg Wielościan”, wyświetlany w oknie rysunku
- Oblicz objętość kształtu alfa za pomocą funkcji „objętość”, która działa dla kształtów alfa
- Przelicz objętość na mm^3
- Wydrukuj objętość bryły w oknie poleceń
- Pytaj o ustawienie nazwiska pacjenta jako danych wejściowych
- Uzyskaj aktualną datę i godzinę z zegarem i ustaw „DateandTime”
- Wywołaj funkcję „patientFiles”, aby zarejestrować i wykreślić obliczone dane
Stąd drugi i trzeci obraz powinny pokazywać wyskakujące liczby, a czwarty obraz, który powinien być wyświetlany w oknie poleceń.
Zalecana:
Wskaźnik głośności Neopikselowy pierścień LED Ws2812 i Arduino: 8 kroków

Wskaźnik głośności Neopixel Ws2812 LED Ring i Arduino: W tym samouczku dowiemy się, jak zrobić wskaźnik głośności za pomocą pierścienia LED Neopixel Ws2812 i arduino. Obejrzyj wideo
Pasek głośności LED: 9 kroków (ze zdjęciami)

Pasek głośności LED: Mój warsztat jest zbyt mdły. Pomimo lakierowanych drewnianych desek w stylu lat 80-tych, które pokrywają moje ściany, brakuje w nim zarówno koloru, jak i oczywiście: diod LED. Podobnie często odtwarzam muzykę podczas lutowania elektroniki. To dało mi do myślenia, czy mógłbym połączyć zarówno muzykę, jak i diody LED
COVID-19 Śledzenie w czasie rzeczywistym dla ESP32: 3 kroki

COVID-19 Realtime Tracker dla ESP32: Ten mały tracker pomoże Ci być na bieżąco z epidemią koronawirusa i sytuacją w Twoim kraju. Wyświetlacz pokazuje naprzemiennie aktualne dane z różnych wybranych przez Ciebie krajów.Dane są zbierane przez stronę internetową www.wo
Śledzenie GPS w czasie rzeczywistym: 3 kroki
Śledzenie GPS w czasie rzeczywistym: W tym samouczku pokażemy, jak śledzić lokalizację w czasie rzeczywistym za pomocą BerryGPS-GSM, Raspberry Pi Zero i stanu początkowego. Wyślemy długość, szerokość geograficzną i prędkość przez 3G z BerryGPS-GSM do stanu początkowego
Robot sieci neuronowej Arduino: 21 kroków (ze zdjęciami)

Arduino Neural Network Robot: Ta instrukcja opiera się na serii 3 części, którą stworzyłem dla kanału Make YouTube, która pokazuje dokładnie, jak prototypować, projektować, składać i programować własnego robota sieci neuronowej Arduino. Po obejrzeniu całej serii powinieneś mieć zakład
