Spisu treści:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:27.
- Ostatnio zmodyfikowany 2025-01-23 15:02.
Autor: Phuc Lam, Paul Yeung, Eric Reyes
Uznanie, że błędy w segmentacji płuc dadzą fałszywe informacje dotyczące identyfikacji obszaru chorobowego i mogą bezpośrednio wpłynąć na proces diagnozy. Nowoczesne techniki wspomagania komputerowego nie zapewniały dokładnych wyników, gdy choroby płuc mają trudne kształty. Te nieprawidłowe kształty mogą być spowodowane wysiękiem opłucnowym, konsolidacją itp. Stosując technikę segmentacji płuc, w której granice płuc są izolowane od otaczającej tkanki klatki piersiowej, nasza aplikacja może zidentyfikować granice z progami wejściowymi użytkownika, aby uzyskać w pełni konfigurowalne widoki kształtów płuc, Celem tego projektu MatLab jest stworzenie przyjaznej dla użytkownika interaktywnej aplikacji do segmentacji płuc w celu wykrywania patologicznych stanów na zdjęciach rentgenowskich płuc. Naszym celem jest stworzenie bardziej skutecznego sposobu ilustrowania i identyfikowania nieprawidłowych płuc, aby dać lekarzom i radiologom bardziej niezawodny sposób diagnozowania chorób płuc. Korzystając z narzędzia do projektowania aplikacji w MatLab, program jest przeznaczony do pracy w szczególności ze skanami RTG klatki piersiowej i tomografii komputerowej (CT), ale jest również testowany pod kątem pracy ze skanami MRI.
Poniższe instrukcje zawierają naszą technikę filtrowania szumu (dolnoprzepustowy filtr Wienera), a także próg obrazu (przy użyciu histogramu intensywności obrazu w skali szarości) oraz przy użyciu gradientu morfologicznego (różnica między dylatacją a erozją obrazu) do zidentyfikować region zainteresowania. Instrukcja wyjaśni następnie, w jaki sposób integrujemy wszystkie elementy w graficznym interfejsie użytkownika (GUI).
Notatka:
1). Ten projekt jest inspirowany artykułem badawczym: „Segmentacja i analiza obrazu nieprawidłowych płuc w tomografii komputerowej: obecne podejścia, wyzwania i przyszłe trendy”. Które można znaleźć tutaj
2). Korzystamy ze zdjęć rentgenowskich z NIH: Clinical Center. Link można znaleźć tutaj
3). Pomoc dla projektanta aplikacji można znaleźć tutaj
4). Przed uruchomieniem kodu: należy zmienić ścieżkę Dir (w linii 34) na katalog plików i typ obrazu (w linii 35) (analizujemy *.png).
Krok 1: Krok 1: Ładowanie obrazu
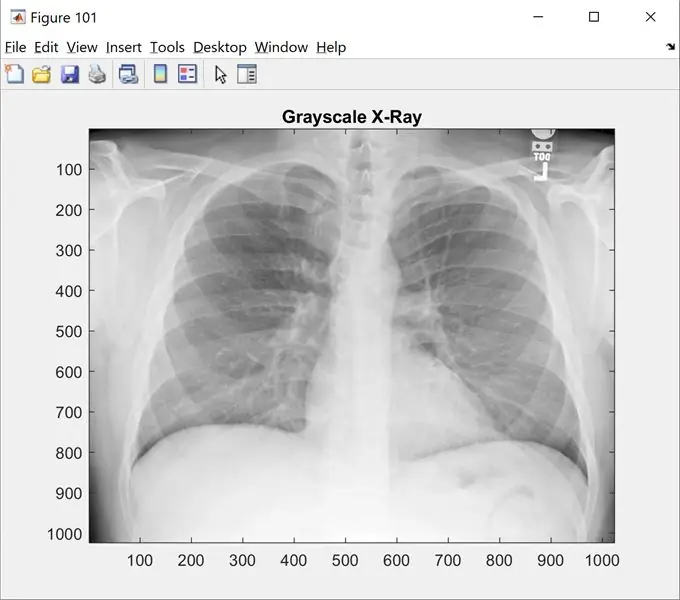
Ten krok pokaże oryginalny obraz w skali szarości. Zmień 'name_of_picture.png' na nazwę swojego obrazu
jasne; kl; zamknij wszystko;
%% Ładowanie obrazów
raw_x_ray='nazwa_obrazu.png';
I=imread(raw_x_ray);
figura(101);
imshow(I);
mapa kolorów (szary);
title('Rentgen w skali szarości');
Krok 2: Krok 2: Filtrowanie szumów i histogram
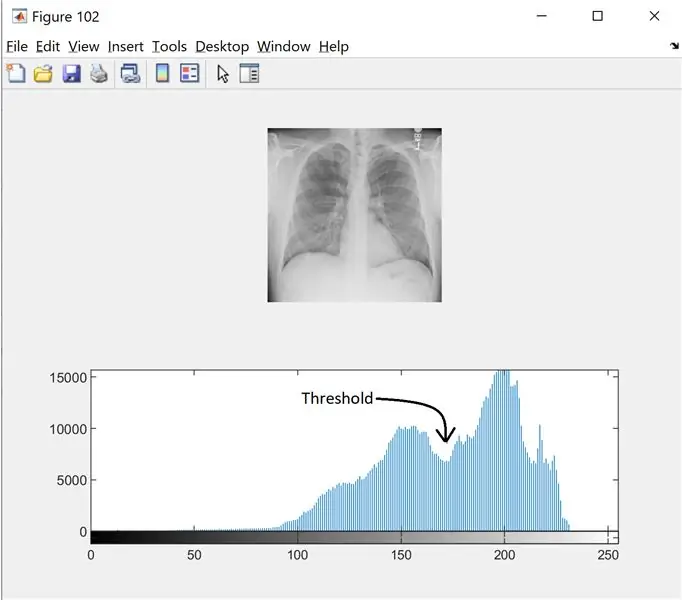
Aby znaleźć próg dla obrazu w skali szarości, patrzymy na histogram, aby zobaczyć, czy istnieją różne tryby. Przeczytaj więcej tutaj
I=wiener2(I, [5 5]);
figura(102);
działka podrzędna(2, 1, 1);
imshow(I);
działka podrzędna(2, 1, 2);
imista (I, 256);
Krok 3: Krok 3: Ustawianie progów
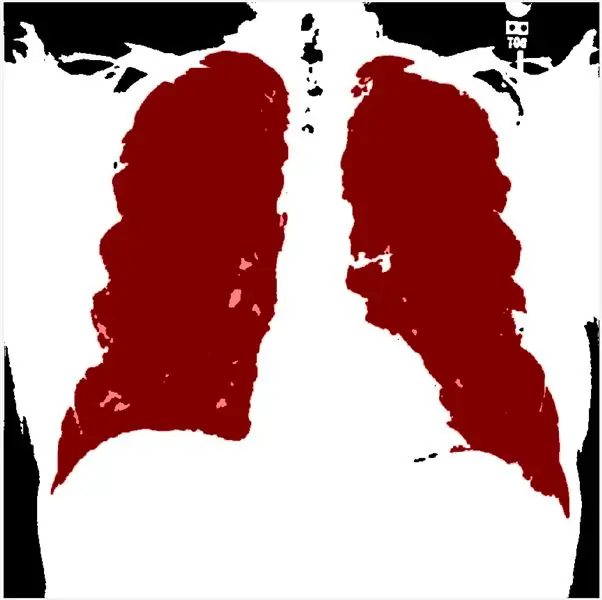
Ten krok pozwala ustawić próg zgodnie z histogramem. morphologicalGradient podświetli interesujący obszar na czerwono, a funkcja visboundaries nałoży obrysowany i przefiltrowany obraz płuca na czerwono.
Używając regionprops, możemy określić tablice solidności i posortować je malejąco. Następnie zbinaryzuję obraz grey sclae i zastosuję metodę gradientu morfologicznego oraz mLoren Shurasking, aby podświetlić interesujący obszar (ROI). Następnym krokiem jest odwrócenie obrazu, tak aby obszar ROI płuc był biały na czarnym tle. Używam funkcji showMaskAsOverlay do wyświetlenia 2 masek. Uwaga: kod jest inspirowany przez Loren Shure, link.
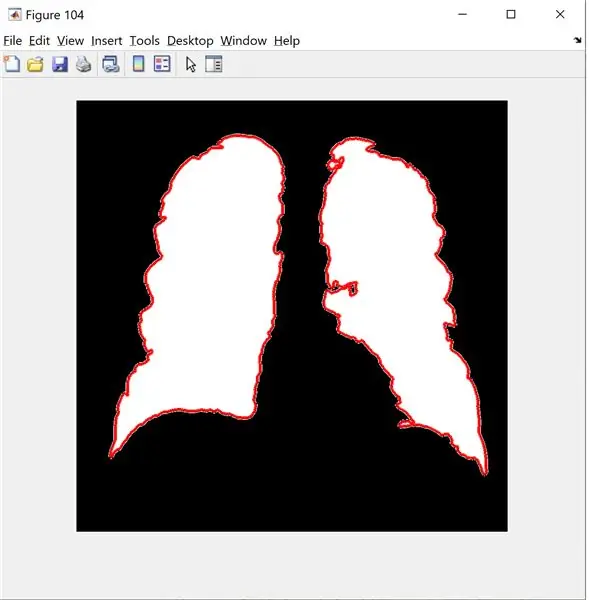
Na koniec tworzę czerwony kontur, używając bwbwboundaries i maskując obraz filtra i granice.
a_thresh = I >= 172; % ustaw ten próg
[labelImage, numberOfBlobs] = bwlabel(a_thresh);
props = regionprops(a_thresh, 'all');
sortedSolidity = sort([props. Solidity], 'descend');
SB = posortowanaSolidność(1);
jeśli SB == 1 % SB akceptuje tylko solidność == 1 odfiltrowuje kości
obraz binarny = imbinaryzacja(I); figura(103);
imshow(obraz binarny); mapa kolorów (szary);
SE = strel('kwadrat', 3);
Gradient morfologiczny = imsubtract(imdilate(binaryImage, SE), imerode(binaryImage, SE));
mask = imbinarize(Gradient morfologiczny, 0,03);
SE = strel('kwadrat', 2);
maska = imclose(maska, SE);
mask = imfill(maska, 'dziury');
maska = bwareafilt(maska, 2); % kontrolny numer obszaru pokaż
notMask = ~maska;
maska = maska | bwpropfilt(notMask, 'Obszar', [-Inf, 5000 - eps(5000)]);
showMaskAsOverlay(0.5, maska, 'r'); % musisz pobrać aplikację/funkcję showMaskAsOverlay
BW2 = imfill(binaryImage, 'dziury');
nowy_obraz = BW2;
nowy_obraz(~maska) = 0; % odwrócone tło i dziury
B=bwgranice(nowy_obraz); % może zaakceptować tylko 2 wymiary
figura(104);
imshow(nowy_obraz);
trzymać się
visgranice(B);
kończyć się
Krok 4: Tworzenie GUI
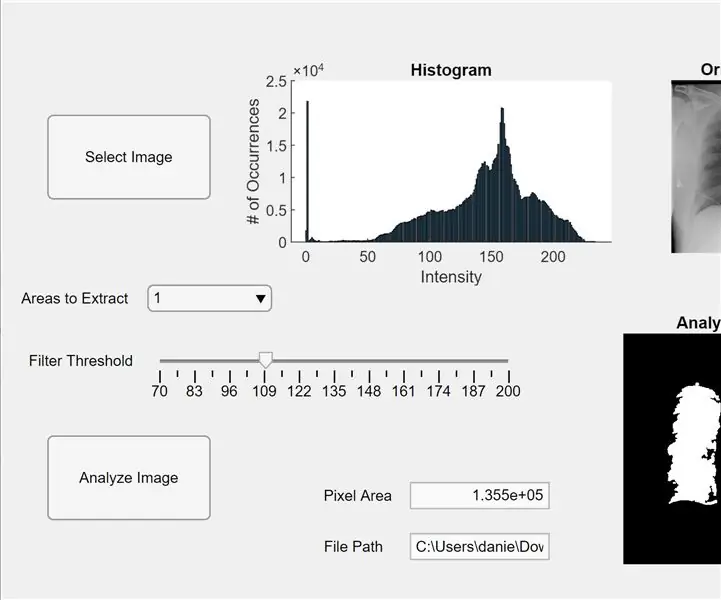
Teraz integrujemy poprzedni kod z aplikacją MATLAB. Otwórz Projektanta aplikacji w MATLAB (Nowy > Aplikacja). Najpierw projektujemy interfejs, przytrzymując i przeciągając w trzech osiach do środkowej przestrzeni roboczej. Następnie klikamy-przytrzymaj-przeciągnij dwa przyciski, jedno pole edycyjne (tekst), jedno pole edycyjne (numeryczne), jeden suwak i jedno menu rozwijane. Każda z dwóch osi wyświetli podgląd i przeanalizowany obraz, a trzecia oś wyświetli histogram pikseli dla „wybranego” obrazu podglądu. Pole edycyjne (tekstowe) wyświetli ścieżkę pliku wybranego obrazu, a pole edycyjne (numeryczne) wyświetli wykryty obszar pikseli w płucach.
Teraz przełącz się z widoku projektu na widok kodu w Projektancie aplikacji. Wprowadź w kodzie kod nieruchomości, klikając czerwony przycisk „Właściwości” ze znakiem plus. Zainicjuj właściwości I, threshold i regionsToExtract, jak w kodzie podanym poniżej. Następnie kliknij prawym przyciskiem myszy przycisk w prawym górnym rogu obszaru roboczego (Przeglądarka komponentów) i przejdź z Callbacks>Go to… callback. Dodaj kod dla „funkcji SelectImageButtonPushed(aplikacja, zdarzenie)”. Ten kod pozwala wybrać obraz do analizy z komputera za pomocą uigetfile. Po wybraniu obrazu pod osiami pojawi się obraz podglądu wraz z histogramem. Następnie kliknij prawym przyciskiem myszy drugi przycisk i powtórz tę samą procedurę, aby utworzyć funkcję zwrotną.
Dodaj kod poniżej „funkcja AnalyzeImageButtonPushed(app, event).” Ten kod wykona zliczanie pikseli i wykrywanie blobów na obrazie podglądu po naciśnięciu przycisku analizy obrazu (w zależności od tego, który został kliknięty prawym przyciskiem myszy dla tego kodu). Po zaprogramowaniu przycisków zaprogramujemy teraz suwak i rozwijane menu. Kliknij suwak prawym przyciskiem myszy, utwórz funkcję wywołania zwrotnego i dodaj kod poniżej „function FilterThresholdSliderValueChanged(app, event)” do końca. Pozwala to suwakowi dostosować próg intensywności szarości.
Utwórz funkcję wywołania zwrotnego dla menu rozwijanego i dodaj kod poniżej „funkcja AreastoExtractDropDownValueChanged(app, event)”, aby menu rozwijane mogło modyfikować liczbę obiektów blob wyświetlanych na analizowanych osiach obrazu. Teraz kliknij każdy element w Przeglądarce komponentów i zmień jego właściwości według własnych upodobań, takie jak zmiana nazw elementów, usunięcie osi i zmiana skalowania. Przeciągnij i upuść elementy przeglądarki komponentów w widoku projektu do funkcjonalnego i łatwego do zrozumienia układu. Masz teraz aplikację w MATLAB, która może analizować obrazy płuc pod kątem obszaru pikseli!
właściwości (dostęp = prywatny)I = ; % plik graficzny
próg = 257; %próg dla binaryzacji intensywności szarości
regionyDoWyciągu = 2;
kończyć się
funkcja SelectImageButtonPush (aplikacja, zdarzenie)
clc;Dir = 'C:\Użytkownicy\danie\Pobrane\images_004\images'; %define plik niezmienny "prefiks"
[imageExt, path] = uigetfile('*.png'); % chwyć zmienną część nazwy obrazu
imageName = [Dir fileep imageExt]; %konkatenacja szczepów niezmiennych i zmiennych
app. I = imread(nazwaobrazu); %przeczytaj obrazek
imshow(app. I, 'rodzic', app. UIAxes); % wyświetl obraz
app. FilePathEditField. Value = ścieżka; %wyświetlana ścieżka pliku, z którego pochodzi oryginalny obraz
kończyć się
funkcja AnalyzeImageButtonPush (aplikacja, zdarzenie)
obraz oryginalny = aplikacja. I;
obraz oryginalny = wiener2(app. I, [5 5]); %filtr usuwania kropek
histogram (app. AxesHistogram, app. I, 256); % wyświetl histogram obrazu
a_thresh = obraz oryginalny >= app.threshold; % ustaw ten próg
labelImage = bwlabel(a_thresh);
props = regionprops(a_thresh, 'all');
sortedSolidity = sort([props. Solidity], 'descend');
SB = posortowanaSolidność(1);
jeśli SB == 1 % SB akceptuje tylko solidność ==1 odfiltrowuje kości
SE = strel('kwadrat', 3);
morphologicalGradient = imsubtract(imdilate(labelImage, SE), imerode(labelImage, SE));
mask = imbinarize(Gradient morfologiczny, 0,03);
SE = strel('kwadrat', 2);
maska = imclose(maska, SE);
maska = imfill(maska, 'dziury');
maska = bwareafilt(maska, app.regionsToExtract);
% kontrolny numer obszaru pokaż
notMask = ~maska;
maska = maska | bwpropfilt(notMask, 'Obszar', [-Inf, 5000 - eps(5000)]);
BW2 = imfill(labelImage, 'dziury');
nowy_obraz = BW2;
nowy_obraz(~maska) = 0;
B = bwboundaries(nowy_obraz); % może zaakceptować tylko 2 wymiary imshow(new_image, 'parent', app. UIAxes2);
hold(app. UIAxes2, 'on');
visgranice(B);
set(gca, 'YDir', 'reverse');
lungArea = bwarea(nowy_obraz);
app. PixelAreaEditField. Value = lungArea;
kończyć się
kończyć się
funkcja FilterThresholdSliderValueChanged(aplikacja, zdarzenie)
app.threshold = app. FilterThresholdSlider. Value;
kończyć się
function AreastoExtractDropDownValueChanged(app, event)stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double(stringNumber);
kończyć się
kończyć się
Zalecana:
Udoskonalenia programowania pociągów w MATLAB: 6 kroków

Udoskonalenia programowania pociągów w MATLAB: Cel: Celem tego zaprogramowanego systemu jest spojrzenie na Arduino na małą skalę i zastosowanie kodowania na większą skalę, aby potencjalnie poprawić niektóre funkcje bezpieczeństwa systemów Amtrak Railroad. W tym celu dodaliśmy czujnik wilgotności gleby
Korzystanie z projektanta aplikacji MATLAB z Arduino: 5 kroków

Używanie MATLAB App Designer Z Arduino: MATLAB App Designer to potężne narzędzie, które umożliwia projektowanie graficznych interfejsów użytkownika (GUI) ze wszystkimi funkcjami MATLAB. W tym samouczku stworzymy GUI do sterowania jasnością diody LED łatwe do naśladowania kroki.N
Samouczek próbkowania sygnału analogowego - MATLAB: 8 kroków

Samouczek próbkowania sygnału analogowego | MATLAB: W tym samouczku pokazujemy, czym jest próbkowanie? oraz Jak próbkować sygnał analogowy za pomocą oprogramowania MATLAB
Roomba sterowana przez MATLAB: 5 kroków

Roomba sterowana przez MATLAB: Celem tego projektu jest wykorzystanie MATLAB oraz zmodyfikowanego programowalnego robota iRobot. Nasza grupa połączyła nasze umiejętności kodowania, aby stworzyć skrypt MATLAB, który wykorzystuje wiele funkcji iRobota, w tym czujniki klifu, czujnik zderzaka
Jak wykonać test słuchu dla dorosłych za pomocą MATLAB: 6 kroków

Jak wykonać test słuchu dla dorosłych za pomocą MATLAB: ZASTRZEŻENIE: Nasz test NIE jest diagnostyką medyczną i nie powinien być jako taki używany. Aby dokładnie zmierzyć słuch, skontaktuj się z lekarzem. Korzystając z materiałów, które posiadaliśmy, nasza grupa wykonała badanie słuchu. Nasz test jest przeznaczony wyłącznie dla dorosłych i nastolatków
