Spisu treści:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:31.
- Ostatnio zmodyfikowany 2025-01-23 15:03.
Autorzy: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Ten kod MATLAB to program do wykrywania dokładnego rozmiaru, kształtu i lokalizacji guza znalezionego w skanach MRI mózgu pacjenta. Ten program został pierwotnie zaprojektowany do pracy z wykrywaniem guza w skanach MRI mózgu, ale może być również używany do diagnostyki raka w skanowaniu innych narządów.
Poniższe instrukcje opisują najpierw metody analizy obrazu poprzez filtrowanie i czyszczenie skanu MRI, poprzez binaryzację, filtrowanie mediany i przesuwanie okien. Następnie poinstruuje, jak wyizolować guz za pomocą wstępnie wygenerowanej maski eliptycznej i przefiltrować ją dalej, aby obrysować obwód kształtu guza.
Po wykryciu nowotworu instrukcje opisują dalej, jak włączyć ten program do graficznego interfejsu użytkownika (GUI). W niniejszych instrukcjach zostanie dołączony odpowiedni kod i pliki, które pomogą wyjaśnić, jak działa analiza skanu MRI.
Kilka rzeczy do poznania, pobrania i przygotowania, zanim przejdziesz do tego instruktażowego: 1. Upewnij się, że masz pobraną najnowszą wersję MATLAB. Możesz zainstalować R2018b tutaj:
2. Aby uruchomić ten program, musisz mieć dostęp do plików skanów mózgu MRI. Chociaż niektóre zawsze można znaleźć na obrazach Google, dokładną i dokładną analizę można przeprowadzić na podstawie odpowiednich obrazów różnych warstw skanów mózgu dla każdego pacjenta. Możesz uzyskać dostęp do plików dla 20 różnych pacjentów z glejakiem przed i po leczeniu z tej bazy danych:
3. Przedmiot tego programu i różne metody, które kierują tym projektem są przedstawione w tym artykule badawczym:
Krok 1: Zainicjuj graficzny interfejs użytkownika (GUI)
Pierwszym krokiem byłoby stworzenie i zainicjowanie graficznego interfejsu użytkownika, GUI. Można to zrobić, wpisując guide w oknie poleceń, naciskając enter i tworząc nowy GUI. Po zakończeniu tego kroku możesz rozpocząć tworzenie funkcji, takich jak osie, tekst statyczny, tekst edycji i przyciski, które będą wyświetlane po uruchomieniu programu i interakcji użytkownika. Funkcje te można edytować i manipulować za pomocą inspektora właściwości, jednak najważniejszą cechą, którą należy zmienić podczas tworzenia tych funkcji, jest nazwa znacznika. Ważna jest zmiana nazwy Tagu każdej implementowanej funkcji, ponieważ pozwoli nam to na stworzenie rozpoznawalnej funkcji zwrotnej. Gdy jesteś zadowolony z układu swojego GUI, możesz przejść do ładowania plików DICOM, które będą wyświetlane w GUI.
Krok 2: Ładowanie i odczyt obrazów MRI w MATLAB
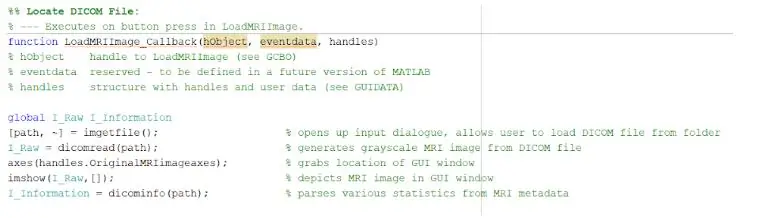
Aby załadować pliki DICOM, należałoby poprawnie zainicjować funkcję oddzwaniania, która byłaby wykonywana po naciśnięciu przycisku „Załaduj obraz MRI”. Po zakończeniu należy utworzyć zmienną globalną, która wyświetli obraz na osiach uchwytów, w których ma być wyświetlany oryginalny obraz MRI. Wszystkie obrazy skanów MRI pobrane z bazy danych to pliki w formacie DICOM, które należy załadować do katalogu MATLAB. Zlokalizuj plik za pomocą imgetfile, aby załadować je do programu. Obrazy są odczytywane za pomocą wbudowanej funkcji MATLAB „dicomread”, a pierwszy surowy obraz dla każdego pliku jest osadzony w lewych osiach GUI za pomocą imshow.
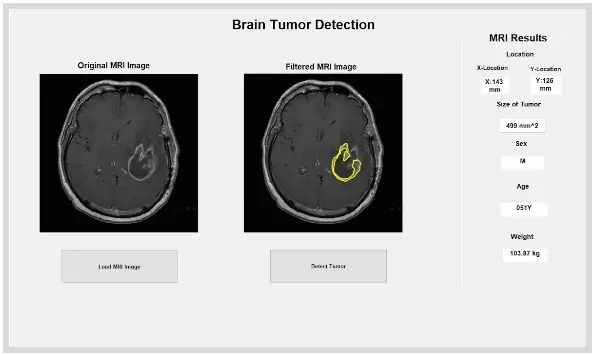
Wbudowana funkcja MATLAB „dicominfo” jest również niezwykle przydatna w adresowaniu wszystkich informacji w każdym pliku dicom MRI. Wykorzystaliśmy tę funkcję, aby wyodrębnić wszystkie informacje opisowe pacjentów, takie jak ich płeć, wiek, waga i wzrost. Ta funkcja zapewnia również kolejność stosów, co jest przydatne do implementacji programu w graficznym interfejsie użytkownika. Stworzyliśmy zmienne dla każdej z opisowych informacji o pacjentach, które będą używane w GUI po naciśnięciu przycisku wykrywania.
Krok 3: Filtrowanie obrazu

Po załadowaniu i odczytaniu pliku DICOM obrazu surowego obraz musi zostać przekonwertowany ze skali szarości do postaci zbinaryzowanej składającej się tylko z czarno-białych pikseli Użyliśmy funkcji 'imbinarize', aby utworzyć obraz binarny z obrazu surowego przez kontrolowanie aspektów adaptacyjnego progowania przy wartości czułości 0,59. Domyślny współczynnik czułości progu 0,5 był niski i nie był w stanie wykryć jaśniejszych plamek i plam z obrazu, więc zwiększyliśmy go do 0,59.
Zbinaryzowany obraz jest następnie przetwarzany przez filtr medianowy przy użyciu funkcji „medfilt2”, ponieważ zbinaryzowany obraz jest dwuwymiarowy. Ustawiamy każdy piksel wyjściowy tak, aby zawierał medianę w sąsiedztwie 5 x 5 wokół odpowiedniego piksela w wejściowym zbinaryzowanym obrazie. Zmniejsza to szum i zachowuje krawędzie w kwadracie 5 x 5 wokół każdego piksela. Następnie stosujemy przesuwane okno za pomocą „strel”, aby utworzyć płaski element strukturyzujący w kształcie dysku o promieniu sąsiedztwa równym 2, aby zidentyfikować każdy centralny piksel początkowy w każdym sąsiedztwie dysku. Wykorzystaliśmy element strukturyzujący dysk, ponieważ analizujemy każdy okrągły punkt i piksele w każdym miejscu, więc element kształtu dysku jest bardziej przydatny.
Po przefiltrowaniu obrazu można go wyczyścić za pomocą funkcji „imclose”, aby usunąć czarne plamy między przefiltrowanymi białymi pikselami na obrazie i zamknąć wszystkie luki wokół niego. Całkowicie przetworzony obraz można następnie wykreślić na drugim wykresie cząstkowym wstępnie przydzielonej figury, umożliwiając porównanie między obrazem surowym i filtrowanym.
Krok 4: Izolacja guza przez maskę eliptyczną


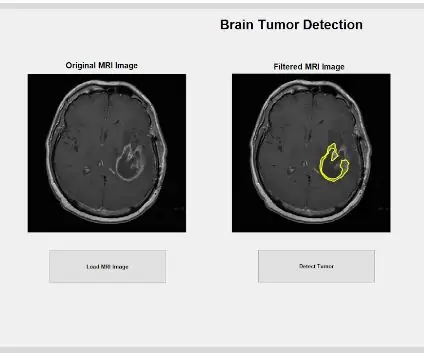
Jasne plamy guza można następnie wyizolować z głównego filtrowanego obrazu za pomocą wstępnie wygenerowanej maski eliptycznej. Aby utworzyć tę maskę, należy znać rozmiar oryginalnego, surowego obrazu skanu MRI i używając długości wiersza i kolumny, odpowiednio jako współrzędnych x i y, przydzielić współrzędne środka eliptycznej. Ustawiamy oś y jako oś główną o promieniu 50 jednostek od środka, a oś mniejszą o promieniu 40 jednostek od środka.
Użyliśmy funkcji MATLAB 'meshgrid' do wygenerowania płaszczyzny kartezjańskiej z dwuwymiarowymi współrzędnymi siatki na podstawie współrzędnych zawartych w wektorach od 1 do długości osi x i od 1 do długości osi y obrazu. Col jest macierzą, w której każdy wiersz jest kopią osi x, a wiersz jest macierzą, w której każda kolumna jest kopią osi y. Siatka kartezjańska reprezentowana przez współrzędne Col i Row ma wiersze length(1:Y_Size) i kolumny length(1:X_Size). Użyj wskaźników Col i Row wygenerowanych przez siatkę kartezjańską, aby określić równanie elipsy w zależności od wcześniej określonego promienia i współrzędnych środka. Eliptyczny kontur można teraz wypełnić białymi pikselami znalezionymi w miejscach guza.
Wykorzystując wstępnie wygenerowaną maskę eliptyczną, możemy wyciąć z przefiltrowanego obrazu konkretny guz, który chcesz analizować. Maska eliptyczna wykrywa, które plamki logicznie mieszczą się w obrysie elipsy i akceptuje to jako plamkę na filtrowanym obrazie jako akceptowalną jako guz. Funkcja „bwareafilt” następnie odfiltrowuje z obrazu wszystkie inne obiekty poza wykrytym guzem. Użyliśmy konkretnego okna o wymiarach 500 na 4000 empirycznie na podstawie wymiarów wszystkich obrazów. Następnie zastosowaliśmy kolejne przesuwane okno ze „strelem” jako elementem strukturyzującym w kształcie płaskiego dysku o większym promieniu sąsiedztwa wynoszącym 6, aby zamknąć luki między każdym centralnym białym pikselem w wykrytym guzie. Wykryta plamka guza jest dodatkowo czyszczona za pomocą „imclose”, aby dodatkowo wyeliminować czarne piksele i wypełnić wszystkie dziury „imfill”. Ten przetworzony guz można następnie wyświetlić na trzecim podwykresie na wcześniej przydzielonym wykresie, aby zapewnić porównanie między wyizolowanym guzem a oryginalnymi i przefiltrowanymi obrazami ze skanu MRI.
Krok 5: zarysowanie guza
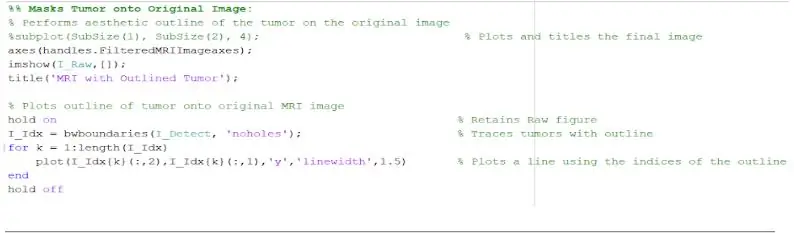
Teraz, gdy guz jest wyizolowany za pomocą maski, można go obrysować i wyświetlić na oryginalnym obrazie, aby pokazać jego dokładną lokalizację. W tym celu użyliśmy funkcji „bwboundaries”, aby prześledzić wcześniej wykryty guz z obrysem. Określiliśmy kontur, aby nie zawierał dziur w obiekcie guza, gdy jest on zarysowany. Można to nanieść na oryginalny, surowy obraz za pomocą pętli „for”, która kreśli kontur wokół guza za pomocą wskaźników linii o szerokości 1,5 piksela. Ten zarys jest następnie nanoszony na surowy obraz, pokazując dokładny rozmiar i lokalizację guza w stosunku do oryginalnego skanu MRI.
Krok 6: Analiza właściwości fizycznych guzów
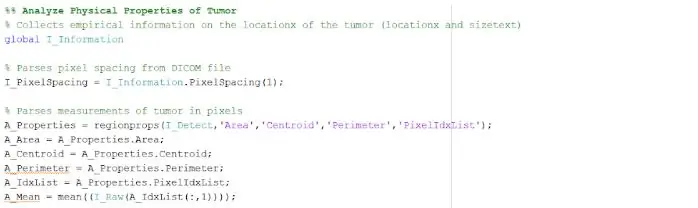
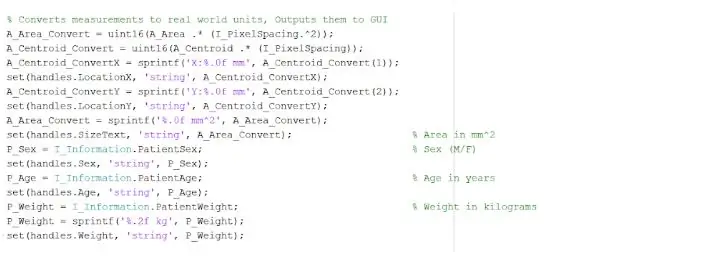
Wyizolowane i zarysowane miejsce może dostarczyć nam przydatnych informacji o wielkości, obszarze i lokalizacji guza. Wykorzystaliśmy funkcję „regionprops”, aby wykryć właściwości guza, które odnoszą się do obszaru, obwodu, centroidów i wartości indeksu pikseli. Ta wartość indeksu pikseli daje nam rzeczywiste jednostki dla każdego piksela każdego obrazu, unikalne dla każdego skanu. Te właściwości można następnie przekonwertować na rzeczywiste jednostki milimetrów. Informacje empiryczne, które dostarcza nam program, są unikalne dla każdego skanu MRI i są niezwykle przydatne w określaniu rozmiaru, lokalizacji i rodzaju guza, które użytkownicy mogą analizować i wprowadzać do graficznego interfejsu użytkownika.
Zalecana:
Wykrywanie wibracji za pomocą modułu czujnika wstrząsu piezoelektrycznego: 6 kroków

Wykrywanie wibracji za pomocą modułu piezoelektrycznego czujnika uderzeniowego: W tym samouczku nauczymy się wykrywać wibracje uderzeniowe za pomocą prostego czujnika piezoelektrycznego Moduł wibracji i Visuino.Obejrzyj film demonstracyjny
Wykrywanie obiektów za pomocą płyt Sipeed MaiX (Kendryte K210): 6 kroków

Wykrywanie obiektów za pomocą płyt Sipeed MaiX (Kendryte K210): Kontynuując mój poprzedni artykuł o rozpoznawaniu obrazów za pomocą płyt Sipeed MaiX, postanowiłem napisać kolejny samouczek, skupiając się na wykrywaniu obiektów. Ostatnio pojawiło się trochę interesującego sprzętu z chipem Kendryte K210, w tym S
Sterowanie Raspberry Pi za pomocą Matlaba: 5 kroków

Kontrolowanie Raspberry Pi za pomocą Matlab: Hej, ten samouczek dotyczy kontrolowania bezgłowego Raspberry Pi za pomocą programu Matlab. Być może będziesz musiał zainstalować matlab do najnowszej wersji, aby uzyskać wsparcie dla nowszej tablicy raspberry pi
Proste wykrywanie kolorów za pomocą OpenCV: 6 kroków
Proste wykrywanie kolorów za pomocą OpenCV: Cześć! Dzisiaj pokażę prostą metodę wykrywania koloru z wideo na żywo za pomocą OpenCV i Pythona. Zasadniczo po prostu przetestuję wymagany kolor w ramce tła lub nie i za pomocą modułów OpenCV zamaskuję ten region i
3 niesamowite projekty kontroli mózgu / umysłu Oświetlenie LedStrip LED z Arduino i Neurosky: 6 kroków (ze zdjęciami)

3 Niesamowite projekty sterowania BRAIN / MIND Oświetlenie LedStrip LED Z Arduino i Neurosky: Czy kiedykolwiek chciałeś włączyć lub wyłączyć światło, myśląc o tym? A może chcesz wiedzieć, jak bardzo jesteś zestresowany, patrząc na kolor diody RGB? Teraz możesz, postępując zgodnie z instrukcjami! Aby poczuć, co zamierzamy
